生体機能物質工学実験 II
第 1 週のレポートへの簡単なコメント
講評:
「分子遺伝学 C」を既に履修した人。講義で学んだ内容をきちんと消化して
“使える知識”
にしているかどうかが重要です。いつでも中味を引き出せるような抽斗に入っていますか?
物置の奥に積んであるだけではだめですよ。
要点:
* VCSM13 を入れたのは、もともと二本鎖 DNA であった pBluescript II SK+ の
一本鎖 DNA を作るためです。覚えていますか?
* 今回の電気泳動で、サイズマーカーを泳動しましたが、サイズマーカーは、あくまで
目安として流しただけです。核酸の電気泳動の場合、同じサイズ(塩基長)の二本鎖核酸
であっても、直鎖状、開環状、閉環状の順に移動度が小さい(泳動が遅い・・・イメージ
できますよね?)ので、直鎖状のサイズマーカーと環状プラスミドとでは、サイズの推定は
できません。
また、同じ長さ(例えば 3 kb)であったとしても、二本鎖と一本鎖では、一本鎖の方が
移動度が大きくなります。もちろん、サイズが半分だからです・・・。ただし、この場合にも、
例えば 3 kb の一本鎖 DNA と 1.5 kb の二本鎖 DNA とで移動度が同じになるかというと、
やはり 3 kb の一本鎖 DNA の方が移動度が小さくなります(これも、何となくイメージでき
ますよね?)。これらを正確に対応づけることは難しく、実際のところサイズの推定は
かなりいい加減にならざるを得ません。
さらに、今回は、一番大きなサイズマーカーは 1857 bp です。プラスミドは、この一番
大きなマーカーのバンドよりも上に位置します。アガロースゲル電気泳動では、アガ
ロースの濃度に応じて、分離可能な核酸のサイズの幅があります。大きすぎたり小さすぎ
たりすると、分子量ごとにきれいに分離されません。つまり、サイズマーカーを利用して
標準曲線を描くとき、分子量を縦軸(対数目盛)に、移動度を横軸にとってグラフを描くと
ある一定の範囲のサイズの核酸のみが直線に乗ることがわかります。したがって、
一番大きなサイズマーカーより大きなバンド、一番小さなサイズマーカーより小さなバンド
については、サイズの推定はできないということになるわけです(下図参照)。
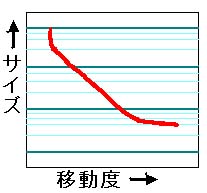
・・・というわけで、今回のサイズマーカーはあくまで目安です。
細かいけれども重要な点:
* 3 M とか、 35 mg/ml というように数字と単位の間にはスペースが必要です。
ただし、75% や 37℃ については、数字の一部という考え方によるのでしょうか、数字と
単位(?)の間にスペースを入れません。来年、卒論で同じことを指摘されないように
いま、しっかりと頭に入れておいてください。
* 遺伝子の名前(lacZ とか dut とか
ung とか)はイタリックです。また、文頭に来ても
大文字にはなりません。一方、タンパク質の名前はイタリックにせず、全部あるいは
頭文字が大文字になることが多いです。ただし、このルールは、違う生物を使っている
人たちの間で必ずしも統一されておらず、厳密なものではありません。
課題 2:
* pBluescript II SK+ は lacZ 遺伝子の翻訳領域の N 末端近くにマルチクローニングサイト
を持っていて、この実習では、そこに EGFP cDNA を組み込んであります。今回の実習
では、lacZ の翻訳の向きと EGFP cDNA の向きは一致していますので、一見、EGFP の
タンパクが発現しそうに思えます。しかし、連結部分の配列をよく見てください。実際に
Web テキストのページから配列を取り出して、自分の手で BamHI
での連結をしてみて
ください。そして、自分の手で、開始メチオニンから “翻訳” してみてください。β-
ガラクトシダーゼの読み枠と、EGFP の読み枠がずれていませんか?これでは、
EGFP タンパクはできません。「β-ガラクトシダーゼとは別に EGFP の部分だけで翻訳
されるのでは?」と思う人もいるかもしれません。しかし、それでは、EGFP cDNA の
開始メチオニンの上流に Shine-Dalgarno
配列はありますか?
これを読んで、「ああ。そうか」と思った人。わかった気にならず、必ず自分の手で一度
配列をきちんとノートや方眼紙に書いて、自分の手で翻訳してください。こういうことは
枝葉末節、取るに足らないことのように思うかも知れませんが、きちんと理解していないと
まともな研究ができるようにはなりません。
* 重要なことがもう一つあります。今回の実習では、SK+EGFP から EGFP タンパクを作る
ことはできませんが、そのことには何の問題もありません。なぜなら pBluescript II SK+ に
組み込んだのは、タンパクを発現させるためではなく、突然変異を導入するために必要な
1 本鎖 DNA を調製するためだからです。タンパクが発現する必要などないのです。
課題 3:
* クロラムフェニコール耐性遺伝子は F' に載っています。クロラムフェニコール存在下で
培養する理由は F' を保持させるためです。 F' に限らず、プラスミドは、大腸菌の生育に
必須のタンパクをコードしているわけではないので、細胞分裂のときに娘細胞に均等に
分配されないなどの理由で、いつの間にかプラスミドを持たない細胞が増えてしまいます。
プラスミドが菌の中にあると、それだけで負担になる(例えば複製に使われる
DNA 合成
酵素をたくさん使ってしまうため大腸菌ゲノムの複製が遅れる?)ので、プラスミドを
持たなくなった大腸菌はよりスピーディーに増殖し、気がつくとプラスミドを持たない菌
ばかりになってしまうのです。したがって、大腸菌にプラスミドを保持させるには、常に
(プラスミドを持っていなければ死ぬ)というプレッシャーを与え続けなければいけません。
F' を持たなければいけない理由は何か?それは、ヘルパーファージである VCSM13
を感染させるためです。VCSM13 は性繊毛上のタンパクに結合して感染します。性
繊毛は、F' 上の遺伝子によって作られるので、大腸菌には、F' を持っていてもらわないと
いけないわけです。
課題 4:
* EGFP cDNA の内部に KpnI の認識配列があったり HindIII の認識配列があると、内部で
切れてしまいますので、cDNA のつなぎ換えがうまくできませんね。同様のことは
pBluescript II SK+ の側にも言えます。
* BamHI と HindIII の組み合わせで cDNA の連結をした場合と、KpnI と HindIII
の組み合わ
せで cDNA を連結した場合とで、pBluescript II SK+ に組み込まれたときの cDNA の方向
は逆になります。しかし、それは問題ではありません。なぜなら、Bluescript を用いて
タンパクの発現実験をするわけではないからです。もちろん、この場合、一本鎖 DNA を
調製する際、今回の実験と相補的な側の鎖が回収されることになりますので、リン酸化
オリゴも、今回用いたのと相補的な鎖の配列で作製する必要がでてきますが・・・。
実験のもくじのページに戻る
