海洋生命・分子工学実験 II
まず,この実習で使う GFP (EGFP)cDNA と,プラスミドの配列を確認しておきます。
配列についての正しい理解が以後の実験に欠かせません。配列情報の中で注目すべき
ところは制限酵素認識配列の位置や数,プロモーターや転写開始点,翻訳開始点,
終止コドンなどです。
知っておかなければならないこと
プラスミドはゲノム DNA と独立に複製する遺伝因子であるということを,「分子遺伝学」
「分子生物学」,「遺伝子工学」などで学びました。プラスミドは,遺伝子を単離したり
切り貼りしたり細胞に導入するために欠かせない道具となっています。そのため,配列に
さまざまな手を加えられた,目的別のプラスミドが多数市販されています。ここで用いる
プラスミドは 2 種類です。一つは大腸菌を用いてタンパクを発現させるために開発された
pQE30
(QIAGEN 社),もう一つは RNA プローブの合成や配列決定,一本鎖 DNA の
調製など多目的に使用される pBluescript II SK+
(Stratagene 社)です。 pQE30 は
大腸菌を用いてタンパクを発現させ,さらに発現させたタンパクを簡単に精製するための
仕掛けが施されています(この実習の後半で恩恵にあずかります)。タンパクの発現には
適していますが,大腸菌細胞 1 個あたりのコピー数が非常に少ないため,プラスミドの精製
や配列決定などで苦労するかもしれません。 pBluescript II SK+ は大腸菌細胞内での
コピー数が非常に多いので,大腸菌からのプラスミドの精製が容易です。
この実習では,pBluescript II SK+ に組み込まれた EGFP cDNA
を切り出して
タンパク発現用の pQE30 に移し替え,EGFP タンパクを発現させます。
今回使うプラスミドは,大腸菌の複製開始点(replication origin)とアンピシリン耐性
遺伝子(ampr)を持っています。これらのプラスミドを導入する大腸菌は,抗生物質
であるアンピシリンの存在下では増殖できずに死んで行きます。従って,大腸菌に
プラスミドを導入する操作をした後,アンピシリンを含む培養液で大腸菌を培養すれば
プラスミドがきちんと導入された大腸菌だけが生き残るという仕組みです。以後,プラスミド
をもつ大腸菌は常にアンピシリンを含む培地でプレッシャーをかけ続けなければいけ
ません。
この実習で使う GFP は正確に言うと野生型の GFP ではなくて,突然変異の入った
ものであり,EGFP と呼ばれています。 EGFP
についての詳しい説明は,別冊実験医学
ザ・プロトコルシリーズ 「non-RI 実験の最新プロトコール(蛍光の原理と実際:
遺伝子解析からバイオイメージングまで)」(編集/栗原靖之・武内恒成・松田洋一)
の第 4 章に載っていますので参考にしてください。
pBluescript II SK+ の配列
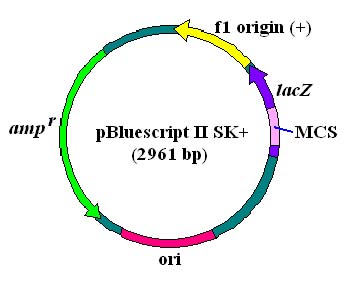
全長 2961 bp のプラスミドです。大腸菌細胞内で複製起点(replication origin
= ori)となる
配列と,β-ラクタマーゼをコードするアンピシリン耐性遺伝子(ampr)があります。その他
気になるのは f1 origin (+)
と書いてある配列です。これが,一本鎖 DNA の合成に重要な
配列なのですが,この実習では使いません。 MCS は multi-cloning site の略で,ここに
たくさんの制限酵素認識配列があり,cDNA や遺伝子断片を組み込めるようになっています。
この部分は,実は lacZ 遺伝子の翻訳領域
N 末端付近にあたります。大腸菌の正常な
lacZ 遺伝子の翻訳領域は全長 3 kb ほどもあるのですが,このプラスミドに組み込まれている
のは正常な lacZ 遺伝子がコードする β-ガラクトシダーゼタンパクの一部分です。このプラ
スミドを導入する大腸菌は,本来もっている lacZ 遺伝子に変異があり,pBluescript II SK+ に
よって持ち込まれる短いタンパクが揃ってはじめて正常な酵素活性を示すようになるのです
(「遺伝子工学」で学んだ α-complementation です)。プラスミドを導入した大腸菌を培養
する寒天培地に合成基質 5-bromo-4-chloro-3-indolyl-galactoside
(X-gal)を含ませて
おくと,できたコロニーが青くなります。これは,基質からガラクトシル基が除去された後の
bromo-chloro-indole
が不溶性の青い沈殿になるためです。ところが,もしも MCS に外来の
DNA が組み込まれると,lacZ 遺伝子のコドンの読み枠(フレーム)がずれたり,途中に終止
コドンができたりします。また,たとえフレームがずれなかったとしても,長い挿入配列によって
lacZ のコードするペプチドの立体構造が異常になるために酵素活性を持たないことになります。
そういうプラスミドが導入された大腸菌のコロニーは青くなりません。プラスミドに外来 DNA を
導入する実験では外来 DNA 導入がうまく行かず,元どおりの pBluescript II SK+ が入った
大腸菌ができてしまう場合があります。こういうとき,プラスミドの導入操作をした後に X-gal を
含む寒天培地に菌をまけば,“白いコロニーを拾う” ことによって正しく組み立てられたプラスミド
の選択を簡単に行うことができます。 MCS 近傍の配列は以下のとおりです。
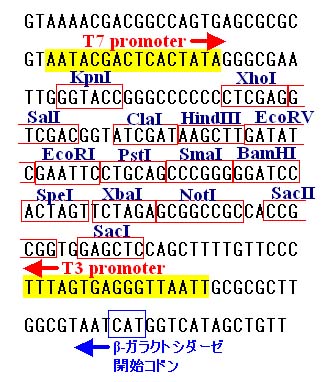
たくさんの制限酵素認識配列が並んでいるのがわかります。四角で囲んだ制限酵素認識配列
は,プラスミド上ではこの図に載っている 1 ケ所しかありません。 HindIII
と BamHI の認識
配列を憶えておいてください。みんなに最初に渡すプラスミドは,この場所に EGFP cDNA が
組み込まれたものです。
pQE30 の配列
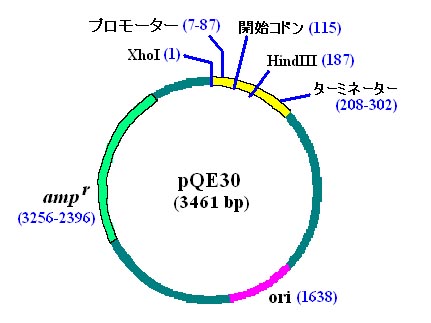
全長 3461 bp のプラスミドです。複製起点(ori)やアンピシリン耐性遺伝子(ampr) をもつ
ところは pBluescript II SK+ と一緒です。このプラスミドには,それらに加えて,組み込んだ
外来 DNA がコードするタンパク質の発現をコントロールする仕掛けが組み込まれています。
その部分の配列を下図に示します。

制限酵素 XhoI の認識配列(CTCGAG)の直後に,バクテリオファージ T5
のプロモーターの
配列があります。プロモーターとオーバーラップして,lac
オペレーターの配列が見られます。
それぞれの配列の役割については「分子遺伝学 C」を復習しておいてください。転写開始点
から
37 塩基下流に RBS という配列(AAAGAGGAG)がありますね。RBS というのは
ribosome-binding
site の頭文字で,別名 Shine-Dalgarno 配列(SD 配列)です。そして
その直後に開始コドンがあります。そのあとにヒスチジン(H)が連続した配列があります。
これについては,この実習の後半で説明します。その後に,制限酵素 BamHI
の認識配列が
あります。そこから,制限酵素 HindIII
の認識配列までの間には,たくさんの制限酵素認識
配列があります。ここが pQE30 の multi-cloning site であり,この制限酵素を利用して
自分の興味のある cDNA などを組み込むのです。最後の HindIII 配列の直後から終止コドン
が 3 つ並んでいます。自分の組み込んだ cDNA が終止コドンを含んでいなくても,ここで必ず
翻訳が終了するようにできています。それに続いて転写終了のシグナルであるターミネーター
配列がありますね。これらの配列を確認しておいてください。
EGFP cDNA の配列
EGFP の翻訳領域の配列は以下のとおりです。
ATG GTG AGC AAG GGC GAG GAG CTG TTC ACC GGG GTG GTG
CCC ATC CTG GTC GAG CTG GAC 60
M V S K G E
E L F T G V V P
I
L V E L D 20
GGC GAC GTA AAC GGC CAC AAG TTC AGC GTG TCC GGC GAG GGC GAG GGC
GAT GCC ACC TAC 120
G D V N G H
K F S V S G E G
E
G D A T Y 40
GGC AAG CTG ACC CTG AAG TTC ATC TGC ACC ACC GGC AAG CTG CCC GTG
CCC TGG CCC ACC 180
G K L T L K
F I C T T G K L
P
V P W P T 60
CTC GTG ACC ACC CTG ACC TAC GGC GTG CAG TGC TTC AGC CGC TAC CCC
GAC CAC ATG AAG 240
L V T T L T
Y G V Q C F S R
Y
P D H M K 80
CAG CAC GAC TTC TTC AAG TCC GCC ATG CCC GAA GGC TAC GTC CAG GAG
CGC ACC ATC TTC 300
Q H D F F K
S A M P E G Y V
Q
E R T I F 100
TTC AAG GAC GAC GGC AAC TAC AAG ACC CGC GCC GAG GTG AAG TTC GAG
GGC GAC ACC CTG 360
F K D D G N
Y K T R A E V K
F
E G D T L 120
GTG AAC CGC ATC GAG CTG AAG GGC ATC GAC TTC AAG GAG GAC GGC AAC
ATC CTG GGG CAC 420
V N R I E L
K G I D F K E D
G
N I L G H 140
AAG CTG GAG TAC AAC TAC AAC AGC CAC AAC GTC TAT ATC ATG GCC GAC
AAG CAG AAG AAC 480
K L E Y N Y
N S H N V Y I M
A
D K Q K N 160
GGC ATC AAG GTG AAC TTC AAG ATC CGC CAC AAC ATC GAG GAC GGC AGC
GTG CAG CTC GCC 540
G I K V N F
K I R H N I E D
G
S V Q L A 180
GAC CAC TAC CAG CAG AAC ACC CCC ATC GGC GAC GGC CCC GTG CTG CTG
CCC GAC AAC CAC 600
D H Y Q Q N
T P I G D G P V
L
L P D N H 200
TAC CTG AGC ACC CAG TCC GCC CTG AGC AAA GAC CCC AAC GAG AAG CGC
GAT CAC ATG GTC 660
Y L S T Q S
A L S K D P N E
K
R D H M V 220
CTG CTG GAG TTC GTG ACC GCC GCC GGG ATC ACT CTC GGC ATG GAC GAG
CTG TAC AAG TAA 720
L L E F V T
A A G I T L G M
D
E L Y K * 239
開始コドンから終止コドンまで 720 bp,239 アミノ酸のタンパクをコードしています。L65 と T66
は,野生型 GFP ではそれぞれ F と S です。このミスセンス突然変異により,EGFP は
野生型 GFP よりも明るい蛍光を発するようになっています。この実習では,みなさんに,まず
EGFP cDNA を組み込んだ pBluescript II SK+ を渡します。そこから作業が開始します。
EGFP と pBluescript II SK+ との連結部分は(ある実験・・・今回の実習とは関係ありません・・・
の目的上)ちょっと複雑な形をしています。その配列は以下のとおりです。
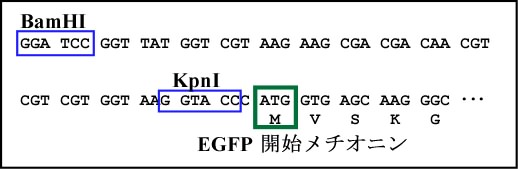
BamHI 部位から EGFP の第一メチオニンまでの間に短い挿入配列があります。この配列に
ついては気にしないでください。一方,EGFP cDNA の後ろ側の配列は,終止コドンより下流
の 3' 非翻訳領域(トレイラーセグメント)で,pBluescript II SK+ の HindIII 配列に連結
されています。終止コドンの後ろから HindIII による切断部位までで 502 bp
あります。
従って,BamHI と HindIII によって切り出される
DNA 断片は 1275 bp です。このことは
後で必要になるのでよく憶えておいてください。
遺伝子工学的実験法の目次へ戻る




