海洋生命・分子工学実験 II
まず,この実習で使うプラスミドの配列を確認しておきましょう。
配列についての正しい理解が以後の実験に欠かせません。配列情報の中で注目すべき
ところは制限酵素認識配列の位置や数,プロモーターや転写開始点,翻訳開始点,
終止コドンなどです。
知っておかなければならないこと
プラスミドはゲノム DNA と独立に複製する遺伝因子であるということを,「分子遺伝学」
「分子生物学」,「遺伝子工学」などで学びましたね? プラスミドは,遺伝子を単離したり
切り貼りしたり細胞に導入するために欠かせない道具となっています。そのため,配列に
さまざまな手を加えられた,目的別のプラスミドが多数市販されています。外来の遺伝子や
cDNA (まとめて外来 DNA と呼びましょう) を組み込むための “乗り物” として使われる
プラスミドを総称して,プラスミドベクター (plasmid vector) と言います。この実習で使う
プラスミドベクターは 2 種類あります。一つは pCR II
(Invitrogen 社,図 1),もう一つは
pBluescript II SK+
(Stratagene 社,図 5)です。pCR II は,もともと,PCR 産物を効率
よくクローニングするために開発されたベクターですが,この実習ではそのことは関係あり
ません。深く考えないことにしましょう。 pBluescript II SK+ は,一本鎖 DNA の調製や
塩基配列の決定,RNA プローブの合成など幅広い用途にとても便利なプラスミドです。
どちらのベクターも,外来の遺伝子や cDNA などを組み込むためにマルチクローニング
サイト (multi-cloning site = MCS) を持っています(図 2 と 図 6 参照)。 MCS には
多数の制限酵素認識配列が所狭しと並んでいます。それらの制限酵素の配列を利用して
その位置に外来 DNA を組み込むことができます。 MCS は,lacZα と呼ばれる遺伝子
(大腸菌のラクトースオペロンを構成する lacZ 遺伝子の N 末端領域をコードする短い
遺伝子) の,翻訳領域の内部にあります。このようなプラスミドを導入するときには,
lacZ 遺伝子に突然変異を持ち,N 末端部分をもたない β-ガラクトシダーゼを発現する
ような大腸菌を使います。そうすると,ベクターの MCS 内に外来 DNA が挿入されて
いない場合にのみ,ベクター上の lacZα 遺伝子から合成される N 末端部分と,大腸菌
自身の lacZ 遺伝子から合成される C 末端側が揃うので,β-ガラクトシダーゼが活性化
できます (これが α complementation です)。 しかし,もしベクターの MCS 内にある程度
以上の長さの外来 DNA が挿入されると,lacZα 遺伝子が正常に機能するタンパクを発現
しなくなります。短い DNA が導入された場合でも,その DNA の長さ (塩基対の数) が
3 の倍数でなければ,β-ガラクトシダーゼ (N 末端側) のフレームシフトが起こるので,
機能する β-ガラクトシダーゼは作られません。 β-ガラクトシダーゼ活性は,発色によって
容易に検出することができます。ですから,その方法を使えば,ベクターに外来 DNA を
組み込む操作が成功したか失敗したかを,簡単に見分けることができるわけで,とても
便利なのです。
〜 この段落に書いたことは,ちょっと難しいですね。実習のときに,少し詳しく説明をしよう
と思います。実は,この実習では,この lacZα がとても重要なのです。
どういう風に重要かというと・・・
外来 DNA を組み込む MCS は lacZα の翻訳領域になっています。この lacZα は,当然
のことですが,大腸菌の細胞の中で発現可能なように,プロモーターや翻訳開始点
を備えていなければいけませんね? それらの配列は実際,ちゃんと備わっています。
ということは,lacZα のコドンの読み枠 (フレーム) と一致させるように外来遺伝子を
組み込めば,その外来遺伝子がコードするタンパク質も発現するのです。わかりますか?
さて。。。今回,みんなに渡すプラスミドは 2 種類です。
一つは, pCR II ベクターの MCS に mCherry cDNA を組み込んだプラスミドです。
もう一つは,pBluescript II SK+ の MCS に EGFP cDNA を組み込んだプラスミドです。
この実習では,pCR II に組み込まれた mCherry cDNA
を切り出して,pBluescript
II SK+ に移し替え,mCherry タンパクを発現させてみようと思います。
pCR II ベクターと,みんなが使うプラスミドの配列
まずは,図 1 を見てください。 pCR II ベクター (Invitrogen 社) の簡単なマップです。
図 1: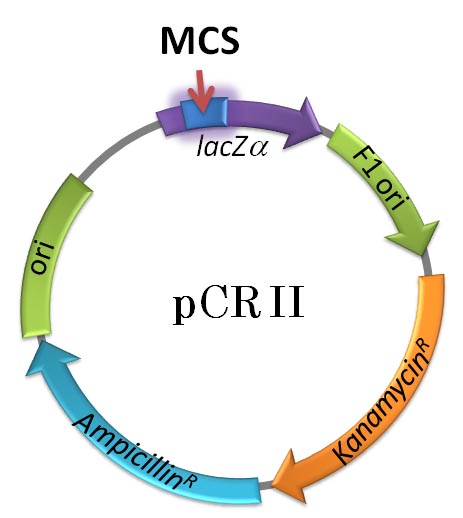
全長 3.9 kb のプラスミドです。大腸菌細胞内で複製起点(replication origin
= ori)となる
配列と,アンピシリン耐性遺伝子 (AmpicillinR…略して ampr とも書きます) および
カナマイシン耐性遺伝子 (KanamycinR) があります。遺伝子は矢印で表されていますが
矢印の方向は転写と翻訳の方向を表しています。今回の実習では後者の遺伝子は
必要ありませんが,アンピシリン耐性遺伝子は重要です。アンピシリンは,抗生物質の
一種です。大腸菌細胞へプラスミドを導入する作業は非常に効率の悪い作業です。それで,
作業が大成功しても,大半の大腸菌にはプラスミドが入りません。そこで,プラスミドを大腸菌
に導入した後は,その大腸菌をアンピシリン存在下で培養します。すると,大腸菌の中に
プラスミドが入っている場合のみ,その大腸菌はアンピシリンを含む培地の上で増殖できます。
プラスミドが導入されなかった大腸菌は,アンピシリン存在下では生存できません。。。
こうして,プラスミドの導入された大腸菌だけが生き残るので,簡単に選別できるというわけ
です。その他,気になるのは F1 ori
と書いてある配列ですが,この実習では使わない配列
なので,とりあえず無視してください。上述のように, MCS にはたくさんの制限酵素認識配列
があり,cDNA や遺伝子断片を組み込めるようになっています (図 2)。四角で囲った制限酵素
認識配列はこのプラスミドベクター (つまり pCR II) には 1 ヶ所しかありません。なお,実際に
Invitrogen 社から買うときには,EcoRI の部分で切断して,末端にちょっとした細工をした状態
で納品されるのですが,今回の実習では関係ないので,そこは省略します。
・・・また,プラスミドは二重らせん型 DNA なのですが,片方の鎖を書いてあれば,相補鎖の
配列は簡単にわかるので,紙に配列を書くときには便宜的に片方しか書きません。また,
lacZα の開始コドンは,青い四角で示してあります。ここまで書けばわかると思う (わかって
ほしい) けれども,図 1 の lacZα の矢印の向き (左から右へ)と,図 2 の配列の向き
(左から右へ) は同じです。
さて,ここでみんなに問題!
(本当は,これも 「当たり前だろ」 と,わかってほしいことなのですが・・・)
【 予習問題 】 図 2 の塩基配列について下記の問いに答えてください。
(1) 図の塩基配列は lacZα 遺伝子の非鋳型鎖でしょうか,それとも鋳型鎖でしょうか?
(2) 図の塩基配列の 5' 末端はどちら側ですか?
(3) lacZα 遺伝子の転写の方向はどちら向きですか?
(4) lacZα 遺伝子がコードする β-ガラクトシダーゼの翻訳の方向はどちら向きですか?
図 2: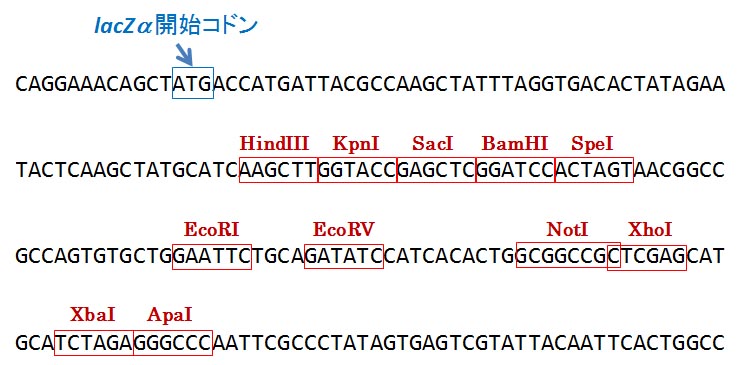
さて,この実習でみんなが使うプラスミドは,“買ったばかりの pCR II そのまま” ではありません。
図 2 の KpnI 配列 (GGTACC) と XbaI 配列 (TCTAGA) の間を切り捨てて,その代わりに
mCherry cDNA を組み込んであります。 図 3 には,ここに組み込んである mCherry cDNA の
塩基配列を載せます。
図 3: 
図 3 では,1 行めの真ん中あたりにある ATG が mCherry の本来の開始コドンです。
また,表示されているアミノ酸配列は,mCherry の読み枠に合わせて翻訳をした場合の
アミノ酸配列です。
さて,賢明な人は既に気づいていることと思いますが,pCR II ベクターの lacZα 遺伝子の
転写/翻訳の方向と,そこに組み込んである mCherry 遺伝子の “本来の” 転写/翻訳の
方向は逆ですよね (図 4)。mCherry cDNA 自体は,ほとんど正味の翻訳領域 (開始コドン
から終止コドンまで) しか備えていません 〜 つまり,独自のプロモーターを持っていません。
したがって pCR II ベクターに組み込まれた mCherry 遺伝子から蛍光タンパク質が生産
されることはありません。 lacZα が転写/翻訳されても,そうしてできる mRNA からは
mCherry のアミノ酸配列を備えたタンパク質は合成されないしね・・・。わかりますか?
図 4: 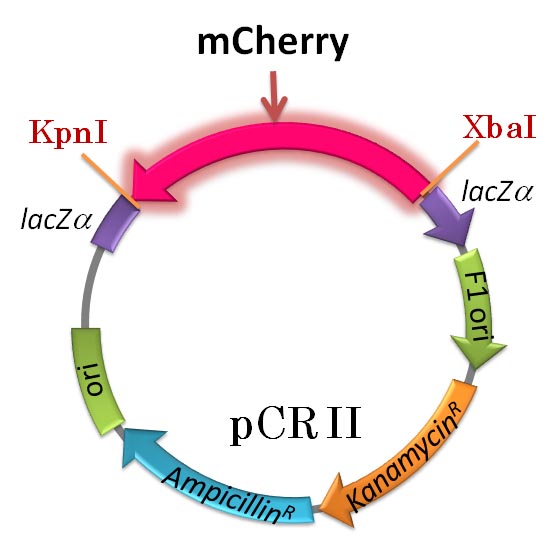
このプラスミドを pCRII-mCherry と呼びましょう。
さて・・・ここでまた問題!
【 予習問題 】 図 2 と図 3 の配列を見て,下記の問いに答えてください。
(5) 図 2 の 2 行めの配列 [ TACTCAAGCTAT・・・ ] に,そのまま続けて pCRII-mCherry の
塩基配列を書いてください。塩基配列は,この問題文に書いた部分を含めて 50 塩基分
を書いてください。
pBluescript II SK+ ベクターと,みんなが使うプラスミドの配列
図 5: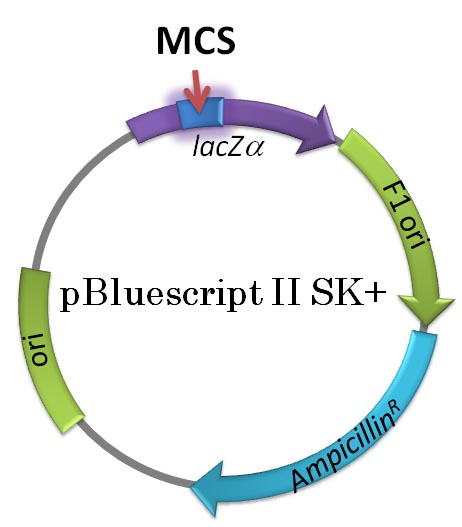
pCR II より若干短い,2.96 kb のプラスミドです。図 1 と見比べてみましょう。カナマイシン
耐性遺伝子を持たないことを除いて,基本的な構造はよく似ていますね。 pBluescript II
SK+ の MCS の塩基配列を図 6 に書きます。
図 6: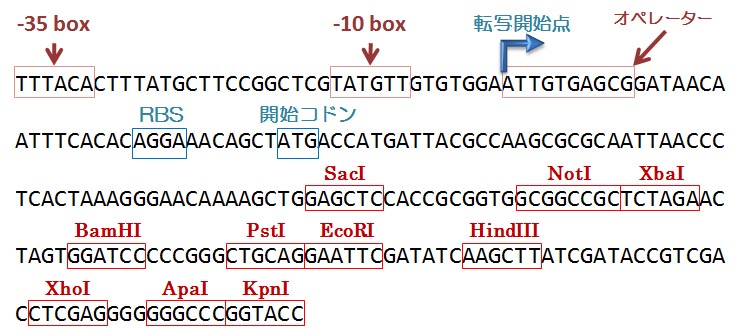
MCS は SacI 配列から KpnI 配列までなのですが,図には lacZα 遺伝子のプロモーター,
オペレーター,リボソーム結合配列 (ribosome-binding site = RBS,別名 Shine-
Dalgarno 配列 = SD 配列),そして lacZα 遺伝子がコードする β-ガラクトシダーゼの
開始コドンも書き込んでおきました。MCS 内には制限酵素認識配列がもっとずっとたくさん
あるのですが,ここではかなり省略してあります (自分で探してみよう)。
さて・・・この実習でみんなに使ってもらうプラスミドは,pBluescript II SK+ ベクターの
そのまんまの形ではありません。図 6 の NotI 配列と XhoI 配列の間を切り捨てて,
そこに EGFP cDNA を組み込んであります。 図 7 には,実際に EGFP cDNA を
MCS に組み込んだ状態の塩基配列を書きます。 図 6 とよ〜く見比べてください。
図 7 の方には SacI 配列から KpnI 配列までしか載せてありません。
図 7: 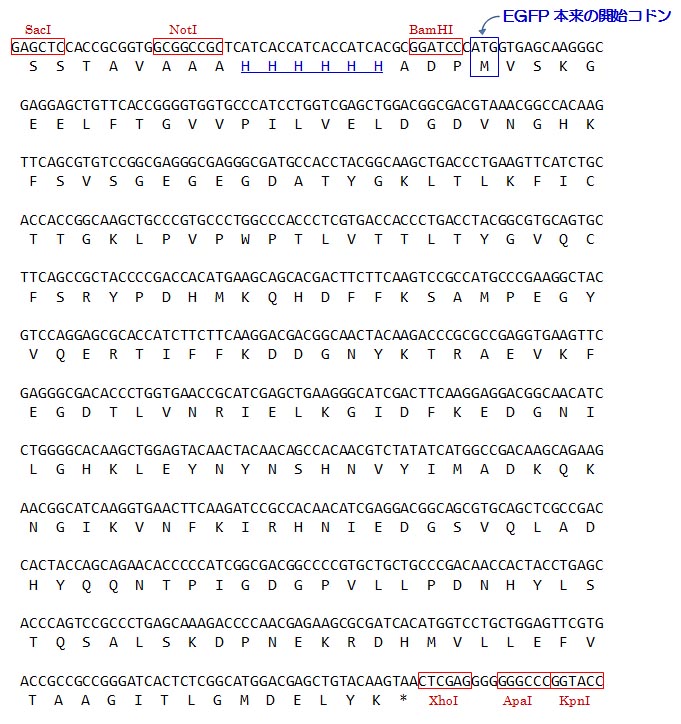
ヒスチジンが 6 個連続した奇妙な配列がありますが,これはある目的のために,人為的に
付加したものです。とりあえず,気にしなくていいです。・・・と言っても,専門海洋生命・
分子工学基礎実験をちゃんと受けた人なら,何のために使う配列か,わかってるよね。
さて,こっちのプラスミドは SK+-EGFP と呼ぶことにしましょう。
ここでも注意してほしいことは,組み込んだ EGFP cDNA には独自のプロモーターが
備わっていないため,lacZα 遺伝子のプロモーターからでなければ,転写/翻訳は
起こらないということです。ですから,EGFP cDNA が “こっち向きに転写/翻訳されて
ほしいなあぁ” という方向と,lacZα の転写/翻訳の方向が一致しなければ,EGFP
タンパクは正常に発現しないのです。
さて・・・ここでまた問題!
【 予習問題 】 図 6 と図 7 の配列を見て,下記の問いに答えてください。問題の中で,
「EGFP の転写や翻訳の方向」 というのは,あくまで正常な蛍光タンパクが発現するために
こっち向きに転写/翻訳されてほしいなあ…という方向のことです
(6) pBluescript II SK+ の lacZα 遺伝子と EGFP cDNA の転写の方向は同じですか?
(7) pBluescript II SK+ の lacZα と EGFP の翻訳の方向は同じですか?
(8) pBluescript II SK+ の lacZα の翻訳領域と EGFP の翻訳領域のコドンの読み枠
は一致していますか? それともずれていますか?
さて,みんなは,この 2 つのプラスミドを使って実習を行います。この先,実験の手順など
を書いたページへ進んで行きますが,このページに書いてあるプラスミドのマップや配列は
常に手元に置いておき,実験中にいつでも見られるようにしておきましょう。また,図に
書き込んでいない情報 (例えば組み込まれている cDNA の長さなど) についても,
自分で調べて,いつでも参照できるようにしておきましょう。
遺伝子工学的実験法の目次へ戻る







