2‐10. 大腸菌を利用した
リコンビナントタンパクの発現誘導
はじめに
遺伝子工学的実験法の 目次のページ に書いたことをもう 1 度見てください。
以下はその抜粋です:
〜生物の体は細胞でできており、”生命活動”の基本は”細胞の機能”であると言えます。
細胞の活動を支える主役は分子機械であるタンパク質です。そして、タンパク質の
設計図が遺伝子です。私たちは、生命活動のメカニズムを知るために、タンパク質の
機能を知る必要があります。そこで、ある特定の機能を探るためには、その機能を担う
タンパク質を直接精製するか、あるいはその設計図を手に入れる必要があります。〜
さて、
これまでの実験で、皆さんはカリガネエガイのヘモグロビンの設計図(cDNA)を単離し、
これをプラスミドに組み込んだわけです。これから、その設計図を利用してヘモグロビン
タンパクを作ってみましょう。ここでは、(転写も翻訳も全て)大腸菌の力を借りて
タンパクを合成し、できたタンパクを電気泳動します。実習ではとりあえずタンパクを
作るだけですが、こういう方法で合成したタンパクは、精製して活性を測定し、機能を
解析することもできるし、正常に機能するタンパクを作ることができればそれを医療など
に応用できるのです。要するに、ここまでの一連の実験で学んだのは、決して実習の
ためだけの初心者専用の練習課題なのではなくて、直ちに第一線に直結する
”役に立つ”技術なのです。そのつもりで、ここまでの過程をよく頭と体に覚え込ませて
おきましょう。
知っておかなければならないこと
皆さんは、十分な説明をされないままで、渡されたプラスミドを使ったわけですが、
ここでプラスミドをもう1度よーく見てください。
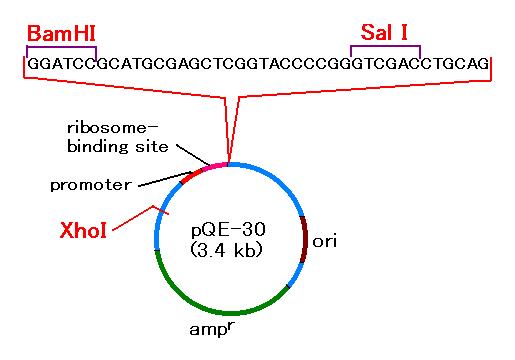
まずは 2-4 で見たプラスミドの図をもう1度見てみましょう(図1)。
図1 pQE-30 プラスミドベクター: ヘモグロビン cDNA
はこのプラスミドの
BamHI 部位と SalI 部位との間に挟み込まれている。

cDNA は、その 5' 側(翻訳されてできるタンパクでいうと N 末端側)を BamHI 側に、
3' 側(タンパクでいうと C 末端側)を SalI 側に向けて組み込まれているのですよね?
(大丈夫?)
cDNA の 5' 側にあたる側のプラスミドの配列には、promoter
(プロモーター)と
ribosome-binding site (リボゾーム結合部位)とがあります。
(「分子遺伝学 C」を思い出そう!)
プロモーターは、RNA 合成酵素の結合部位で、mRNA の転写はここから始まります。
このプラスミドは T5 ファージの強力なプロモーターを持っています。また、
プロモーター(RNA 合成酵素結合部位)のすぐ下流に lac オペレーター
(ラクトースオペロンのリプレッサー結合部位の配列)があります。
(これも「分子遺伝学 C」で教わったはず!)
念のために繰り返しますが、ラクトースオペロンでは、リプレッサーと呼ばれる
タンパク質がオペレーター配列に結合して転写を抑制しています。ラクトース(乳糖)
を分解する酵素群の遺伝子が並ぶラクトースオペロンの場合、 lacI遺伝子が作る
リプレッサーがオペレーターに結合して転写を抑制しています。培地にラクトースを
加えると、リプレッサーはラクトースと結合して立体構造が変化し、オペレーターから
離れます。その結果、抑制が解除してプロモーターからの転写が活性化し、
ラクトース分解酵素群が生産されます。ラクトースの消費によって培地中のラクトースが
減少すると、リプレッサータンパクに結合するラクトースも少なくなります。ラクトースと
離れたリプレッサーは再びオペレーターに結合し、転写を抑制します。そしてラクトース
分解酵素群の生産は停止します。
・・・では、なぜpQE30 の T5 プロモーターにわざわざ lac オペレーターをつけたの
でしょうか?
それは、組み込んだ cDNA からのタンパクの発現をコントロールするためです。
そもそも我々が研究の対象にしようとするタンパクは、何らかの重要な生理活性を
持っています。細胞の機能に大きな影響を及ぼすそのようなタンパクは、往々にして
大腸菌にとっては迷惑な(大腸菌の生育の負担になったり害になるような)タンパク
なわけです。そこで、常時発現させておくと大腸菌が生育せず、結局タンパクを得られ
ないという結果になってしまいます。そこで、タンパクを発現させる実験の場合、まず
組み込んだ遺伝子の発現を抑制した状態で大腸菌をある程度増殖させ、その後に
遺伝子発現を誘導するという方法がとられます。
JM109 系統の大腸菌は F' エピゾームを持っています。F' の中には lacIq
遺伝子があります。 lacIq は野生型の lacI よりも強力なリプレッサーを作る
変異遺伝子です。
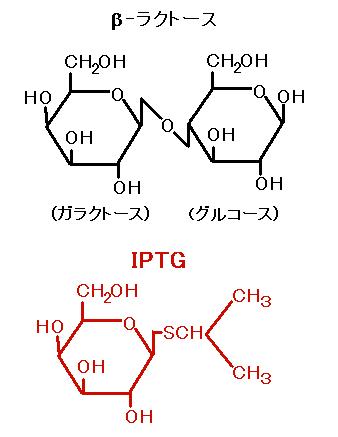
この実験では、ラクトースのかわりに、イソプロピル-β-D-ガラクトピラノシド
(IPTG)を用いて転写を活性化します。IPTG(図2)は、ラクトース同様にリプレッサー
と結合し、オペレーターから引き離しますが、ラクトースと違って分解されて減ることが
ないので、長時間安定に転写活性を維持することができます。その結果、合成される
タンパクは大腸菌の細胞の中で最も量の多いタンパクになります。
図2 ラクトースと IPTG。似てる?
次に、pQE30 のプロモーターから cDNA の連結部分までの配列を見ましょう(図3)。
このプラスミドには転写調節領域(プロモーターとオペレーター)、リボゾーム結合部位、
開始コドンが含まれており、その下流に制限酵素切断部位を持っています。
自分の目的の cDNA をこの制限酵素切断部位に組み込むだけで、 cDNA がコード
するタンパクを発現させることができるようにデザインされているのです。
図3 図の見方は大体わかるはず。T5 プロモーターと lac
オペレーターが
赤字で、制限酵素切断部位を紫で示してある。青矢印で示したのが転写開始点。
ここから mRNA の転写が始まる。RBS はリボゾーム結合部位。開始コドンも
青字で示してある。ここから BamHI 認識部位までがコードするアミノ酸配列
[ MRGSHHHHHHGS ] はプラスミドにコードされている配列。 BamHI が
プラスミドとヘモグロビン cDNA との連結部位である。ヒスチジン(H)が6個
並んでいるがこれに関しては、次の 2-10 で解説する。(プラスミドの配列は
QIAGEN 社の説明書より引用)

図3 に示した塩基配列からわかるように、いま発現させようとしているタンパクは、
バクテリオファージのプロモーターに大腸菌のオペレーターとリボゾーム結合
配列、さらに完全に人工的なヒスチジンリピート、そして軟体動物カリガネエガイの
ヘモグロビンの翻訳領域が組み合わされてできた人工遺伝子の産物です。
このように組換え DNA (recombinant DNA)技術によって作製された遺伝子の
産物をリコンビナントタンパクといいます。
実験 (簡単でごめん)
1. トランスフォーメーション後のプレートから爪楊枝を使って大腸菌の
コロニーをとり、1.5 ml の LB/amp 培地(試験管)に植え、
一晩振盪培養する。
1 人 1 コロニーを、前の晩に教官が用意しておきます。皆さんは
この作業をする必要はありません。
2. いっぱいに増えた大腸菌を全てエッペンに移し、2-7 の実験
(プラスミド DNA の精製)に使う。3. 試験管に残った大腸菌に、1 mM IPTG を含む LB/amp 培養液
を 2 ml 加え、再び 37℃ で振盪培養する。4. 37℃ で 2 時間程度培養した後、試験管の大腸菌を
エッペンに移し、遠心して菌を沈殿させる。5. 上清を捨て、SDS サンプルバッファーを加えてボルテックスで混ぜる。
SDS サンプルバッファーの組成は物質科学実験 CIII のレジュメを参照。6. 80℃ から 90℃ 程度で 5 分煮て、菌を溶かす。
注: 菌が溶けると染色体 DNA が出てきて液がネバネバするので、
ボルテックスをして DNA を断片化して液の粘性を下げておく。7. SDS-PAGE によってタンパクを分離し、クマシーブルー染色をする。
SDS-PAGE に関する資料は別に配ります。