みんなの実験の結果 (2002 年度)
はじめに
このページには、2002 年度の、みんなの実験のデータを載せてあります。
レポート作成用に使ってください。
RNA 抽出
2-1 で抽出した RNA
(100 μl の DEPC処理水に溶かしてある)のうち、 10 μl を
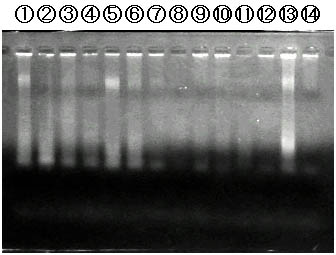
1% アガロースゲルで電気泳動した結果です(図1A, B)。
レポートに使ってください。
うまく行っていれば、28S リボゾーム RNA (約 4.8 kb)、18S リボゾーム RNA
(約 1.9 kb)、トランスファーRNA (約 70 bases)あるいはヘモグロビン mRNA
(約 700 bases)が、バンドとして確認できるはず・・・でしたが・・・。
その他、かなり移動度の小さい(サイズの大きい)位置に明るい部分がありますが、
これは 染色体 DNA の混入です。 AGPC 法では、しばしば DNA の混入が起こります。
失敗というわけではありませんので、落ち込まないように。
さて、皆さん、上で述べたいろいろな種類の RNA が下の写真の
どのバンドに相当するか、わかりますか?
図 1
A 
各レーンのサンプルは以下のとおりです。
1: 野間 2: 真田 3: 江川 4: 長谷川 5: 戸田 6: 土居
7: 山口 8: 石田 9: 板原 10: 花野 11: 岩田 12: 西岡
13: 一戸 14: 北村
B 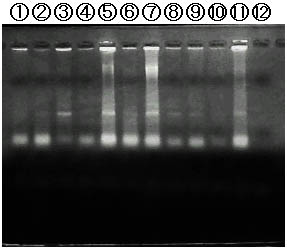
各レーンのサンプルは以下のとおりです。
1: 三田 2: 迫間 3: 太田 4: 市成 5: 渡辺 6: 坂部
7: 平井 8: 竹岡 9: 嶋田 10: 小宮山 11: 松田 12: 河田
* ちょっとだけ不安になってきたので、試しに 3 人分の RNA を使って、実習の
プロトコルどおりの方法で逆転写をした後、ヘモグロビンの cDNA を
PCR で増幅してみました。その結果が下の写真です。
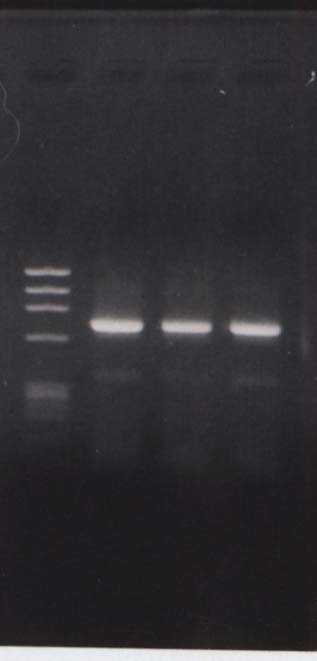
・・・安心した。。。
PCR
2-3 で行った PCR の反応液
50 μl のうち 5 μl 分を電気泳動しました。
その結果が図 2 です。
図 2
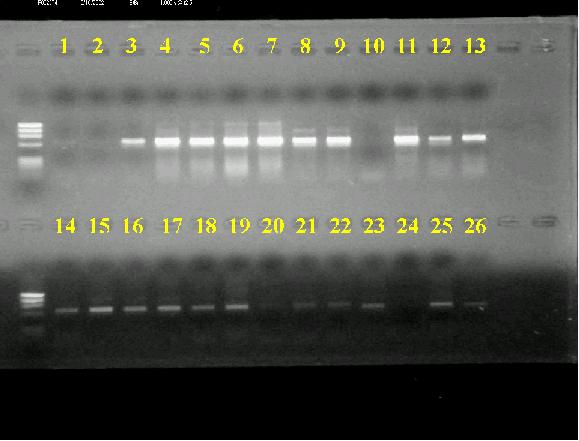
各レーンに流したサンプルは以下のとおりです。
1, 板原; 2, 一戸; 3, 長谷川; 4, 竹岡; 5, 迫間; 6, 渡辺; 7, 松田;
8, 江川; 9, 野間; 10, 土居; 11, 太田; 12, 山口; 13, 平井; 14, 嶋田;
15, 花野; 16, 戸田; 17, 真田; 18, 岩田; 19, 坂部; 20, 北村; 21, 三田;
22, 小宮山; 23, 市成; 24, 河田; 25, 石田; 26, 西岡
なお、サイズマーカーは φX174/HaeIII です。
アガロースゲルからの DNA 断片の精製
2-5 で行った、ゲルからの
DNA 断片の精製の結果です。
皆さん、自分のサンプルが何番目のゲルの何レーンめか、わかりますか?
図 3
A 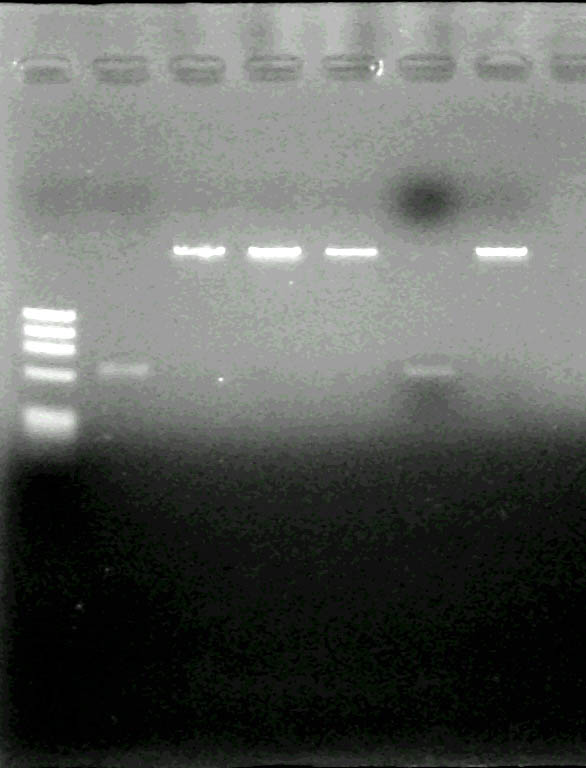 B
B 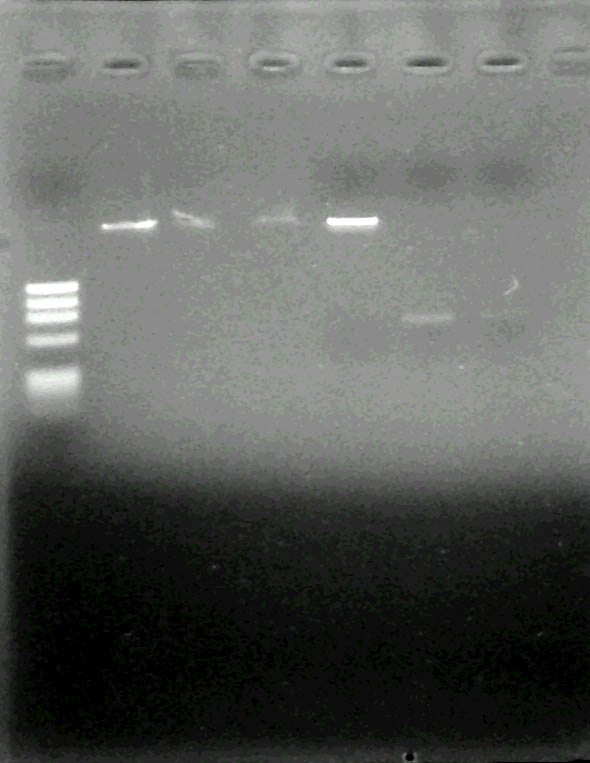
C 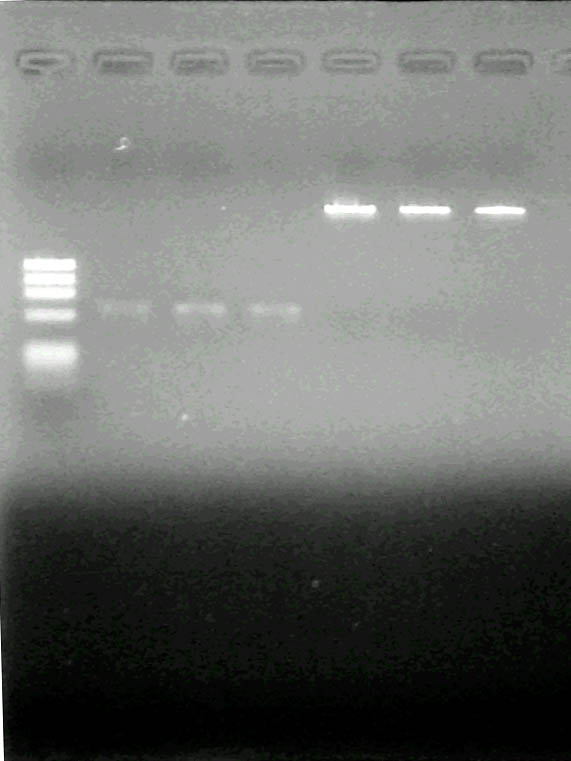 D
D 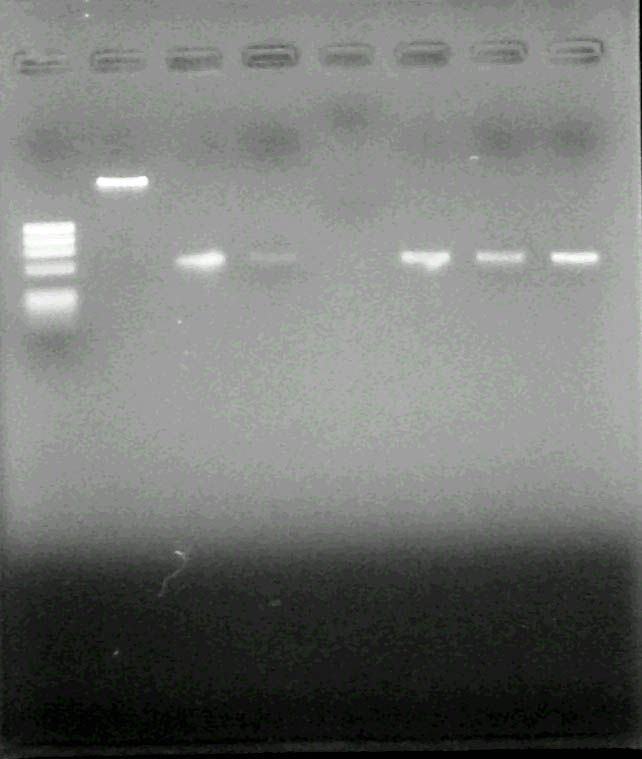
サイズマーカーは φX174/HaeIII です。
大腸菌へのプラスミドの導入
2-6
で行った、トランスフォーメーションの結果の例です。
寒天培地のプレートをそのままスキャナーで取り込んでみましたが、
コントラストをつけてもコロニーは見にくかったので、例としてプレート 1 枚分だけ
ここでお見せします。
通常の実験では、1 枚のプレートに数百個から多いときは数千個のコロニーができる
はずなのですが・・・。実は、みんなのプレートにはコロニーがほとんどできませんでした。
でも、それは皆さんのやり方が悪かったせいではなくて、おそらくプロトコルに問題が
あったものと思っています。いろいろな理由が考えられますが、みなさんに考えてもらう
ことにします。
木曜日に行ったトランスフォーメーションでは、実験の web テキストどおりに JM109
系統の competent cells を使いましたが、全くコロニーができなかったので、試しに
金曜日のトランスフォーメーションでは別の系統(RB791)の competent cells を使い
ました。金曜日の分についてはライゲーションは 30 分ではなく一晩行いました。
図 4

図中の矢じりの先に色が少し濃くなって見える部分がコロニーです。
プラスミドの制限酵素チェック
2-8 で行った、制限酵素チェックの結果です。
図 5
A 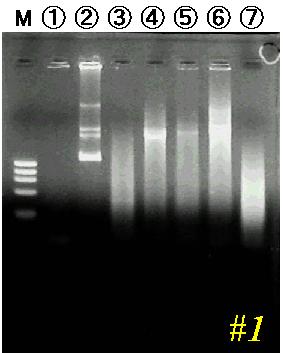 B
B 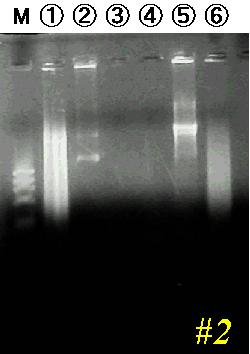
C 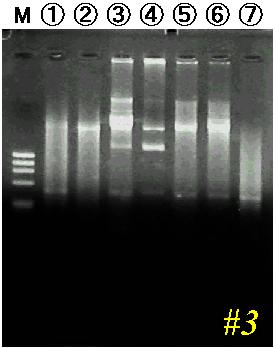 D
D 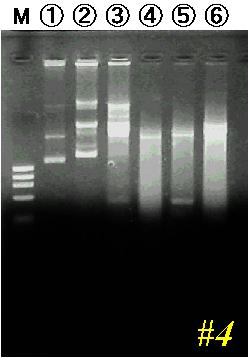
各ゲルのレーン M は DNA サイズマーカー(φX174/HaeIII)です。各レーンのサンプル
の持ち主の名前を書きます。名前の後の ( ) 内に書いてある数字は、その人の
持っているコロニー(クローン)の番号です。ただし、ヘモグロビンが入っていることを
確認済みのプラスミドを持っている人については ( ) 内に BvII と書き、pQE-30 を
持っている人については ( ) 内に QE と書いてあります。
ゲル #1: 1, 岩田(BvII); 2, 竹岡(QE); 3, 太田(8); 4, 市成(5); 5, 西岡(8);
6, 一戸(6); 7, 板原(4)
ゲル #2: 1, 花野(9); 2, 河田(QE); 3, 嶋田(BvII); 4, 坂部(9); 5, 長谷川(7);
6, 野間(2)
ゲル #3: 1, 戸田(2); 2, 江川(7); 3, 真田(1); 4, 平井(BvII); 5, 迫間(10);
6, 松田(4); 7, 渡辺(10)
ゲル #4: 1, 山口(QE); 2, 石田(3); 3, 土居(6); 4, 三田(5); 5, 北村(3);
6, 小宮山(1)
以上です。同じコロニーに由来する、配列の同じクローンを持っている人どうしで
バンドのパターンは一致していますか?
リコンビナントタンパクの発現誘導と SDS-PAGE
2-10 で行った SDS-PAGE の結果です。
図 6
A 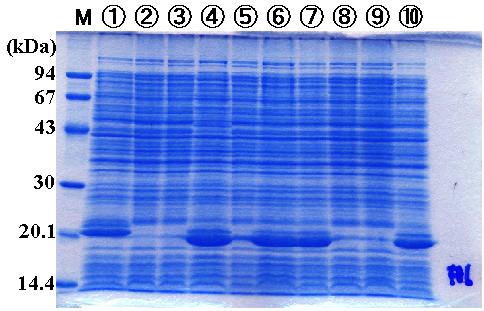
B 
C 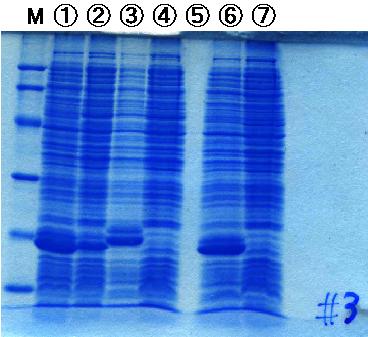
用いたゲルは 15% の SDS-ポリアクリルアミドゲルです。
各ゲルのレーン M はタンパクの分子量マーカー(Pharmacia 社)です。それぞれの
バンドの分子量を A の図に示しています。
本来なら、IPTG を入れる前の大腸菌と IPTG でタンパクの発現を誘導した後の
大腸菌のサンプルを調製して隣のレーンに泳動し、比較することによって誘導された
タンパクを同定するのですが、今回は人数が多いので省略しました。その代わり、
pQE-30 のみのサンプルでは大きな分子量のタンパクはできないはずなので、
これをコントロールとすることができると思います。ゲル上にはたくさんのバンドが
出ていますが、これは、大腸菌をそのまま溶かして泳動用のサンプルにしたためです。
ゲル #3 の 5 レーンめは、サンプルがありませんが、おそらくレーン 5 に流すべき
サンプルをレーン 4 に流してしまったものと思います。サンプルを注ぎ込んだ後の
泳動前のゲルを見て、そんな感じがしました。
さて、ヘモグロビンの cDNA の配列と pQE-30 のプラスミドの配列をよく見て、
期待されるリコンビナントタンパクの分子量を計算してください。その分子量と比べて、
みんなのタンパクの分子量はどうだったでしょうか?
各レーンのサンプルの持ち主の名前を書きます。名前の後の ( ) 内には、図 5 と
同様に、その人の持っているコロニー(クローン)の番号を書いてあります。
ゲル #1: 1, 戸田(2); 2, 江川(7); 3, 真田(1); 4, 平井(BvII); 5, 迫間(10);
6, 松田(4); 7, 渡辺(10); 8, 三田(5); 9, 小宮山(1); 10, 北村(3)
ゲル #2: 1, 市成(5); 2, 長谷川(7); 3, 西岡(8); 4, 一戸(6); 5, 板原(4);
6, 山口(QE); 7, 竹岡(QE); 8, 石田(3); 9, 土居(6)
ゲル #3: 1, 岩田(BvII); 2, 太田(8); 3, 野間(2); 4, 花野(9); 5, 坂部(9);
6, 嶋田(BvII); 7, 河田(QE)
興味深い結果が出ていますね。
リコンビナントタンパクの精製
2-11 で精製したリコンビナントタンパクの SDS-PAGE の結果です。
図 7
A 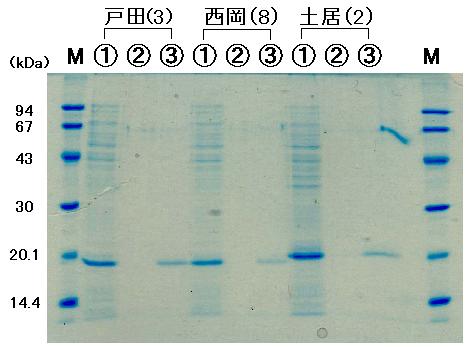
B 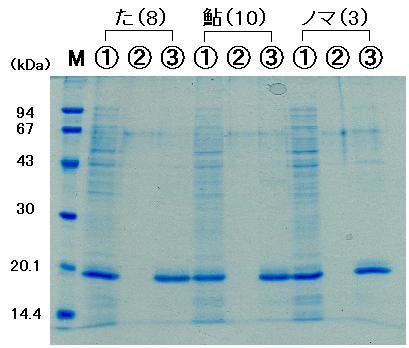
C 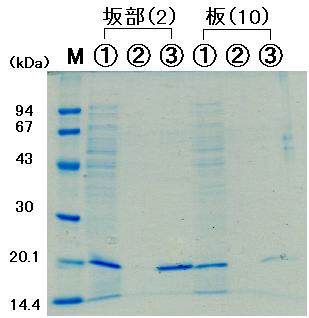
電気泳動の分子量マーカー(M)は前回の SDS-PAGE に使ったものと同じです。
上に書いてある名前の後の ( ) 内の番号は、クローンの番号です。
タンパク発現と制限酵素処理の結果と見比べてください。
各レーンの上に ①、②、③ という番号がついていますが、
①: 大腸菌の溶菌液(遠心後の可溶性タンパク)
②: 1 回めの洗浄の後の洗浄液(バッファー C)
③: バッファー E' で溶出(elute)させた上清(eluate)
です。さて、皆さんの精製の作業はうまくいっていたでしょうか?
塩基配列決定
2-9 で、組み立てたプラスミドの塩基配列決定を行いましたが、残念ながらどの
グループのサンプルでもシグナルが出ませんでした。
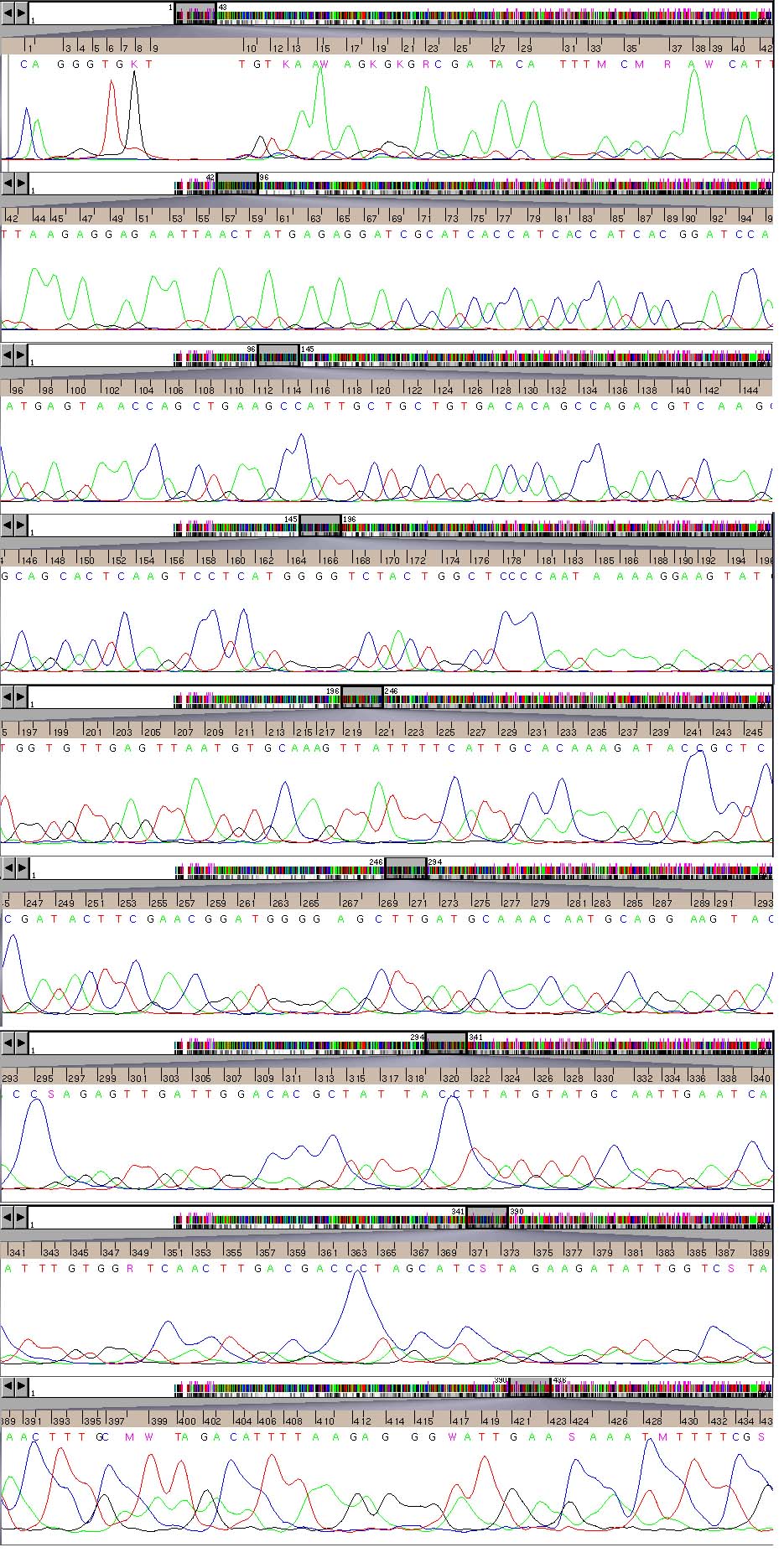
昨年の実習の塩基配列データを載せておきますので、参考にしてください。
わからないことは、生化研の宇田君か、藤原のところまで訊きに来てください。
参考データ
図を小さくすると文字が読めなくなってしまうので、元どおりのサイズで載せています。