2‐11. リコンビナントタンパクの精製
はじめに
この実験で作ったプラスミドは、自分の単離した cDNA がコードするタンパクを簡単に
大量に発現させるためにデザインされたものであることが、2‐9 までの実習でわかった
と思います。しかし、このプラスミドの便利さはまだまだ他にもあるのです。日程の都合上、
皆さんが実際に作業することはできないかも知れませんが、ここで紹介するのは
大腸菌に発現させたリコンビナントタンパクをワンステップで簡単に精製する手法です。
皆さんが前の実験で合成したリコンビナントヘモグロビンタンパクの量は、確かに、
大腸菌が本来持っている全部のタンパクを足したよりも多いくらいでした。しかし、
それでも SDS-PAGE の結果からもわかるように、皆さんがほしいヘモグロビンタンパク
は、(それぞれの量は少ないとはいえ)数え切れない大腸菌タンパクの中に混ざって
存在しているというのも事実です。例えば、皆さんはこのヘモグロビンの酸素結合能を
解析したいとします。そのとき、もし、大腸菌の中に、このヘモグロビンの何十倍、何百倍の
強さで酸素と結合するタンパクがあったとしたら、例え量が少なくてもそのタンパクの存在は
無視できなくなり、実験結果も信用できないものになります。そこで、できることなら、
目的のタンパクだけを精製しなければなりません。今回使った pQE-30 のようなプラスミドは、
作ったタンパクを精製するために便利な工夫がほどこされています。
知っておかなければならないこと
2‐9 で見た配列をもう1度見てみましょう(図1)。前の実験で合成したタンパクは、
正味のヘモグロビンの配列だけでなく、N 末端に余分のアミノ酸配列が付加されて
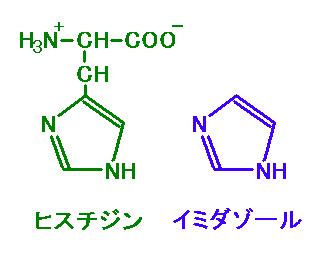
います。特に重要なのは、 6 個ならんだヒスチジンです。
図1 pQE30-BvII の配列

この 6x ヒスチジンリピートが、リコンビナントタンパクを精製するための便利な道具
なのです。図2 を見てください。右側にうぐいす色の壁があります。これは、固相を
あらわしています。実習で使うとすれば、この部分はアガロースゲルです。固相から
飛び出している枝の先(左端)の部分を Nitrilotriacetic acid (NTA)
といいます。
NTA は、ニッケルイオン(Ni2+)の 6 つの結合箇所の
4 箇所を捕まえています。
つまり 2 箇所がフリーです。その残った 2 箇所にヒスチジンが結合するのです。
図では左側の青いジグザグの太線がタンパク質のペプチド骨格をあらわしていて、
そこから 2 個のヒスチジン側鎖が出ている様子を示してあります。
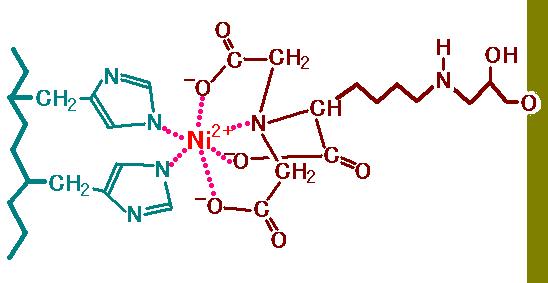
この性質を利用したアフィニティークロマトグラフィーによって、リコンビナントタンパクを
簡単に精製できるのです。N 末端に付加されたヒスチジンリピートは、たくさんの荷物の
中から自分の荷物を見分けて選び出す荷札(タグ)にたとえて、ヒスチジンタグ
(His-タグ)と呼ばれます。
His-タグは、タンパク全体の立体構造を壊すほど大きくないので、多くの場合、
このタグをつけたまま活性の測定などの実験に使うことができます。もしも、タグが
そのタンパクの機能をじゃまするような場合には、タグを C 末端につけるようなことも
できます。
この実験に使った pQE シリーズの他にも、タンパク発現・精製用のプラスミドはたくさん
あります。例えば、Pharmacia から売られている pGEX ベクターは、約
26 kDa の
グルタチオン-S-トランスフェラーゼ(GST)の翻訳領域の下流に自分の
cDNA を
組み込んで、融合タンパクとして発現させるシステムです。この場合、融合タンパクは、
GST がグルタチオンと結合する性質を利用したアフィニティークロマトグラフィーによって
精製することができます。精製したタンパクは融合タンパクのまま活性測定等に使う
こともできますし、GST 部分と切り離すこともできます。GST 翻訳領域の下流、cDNA
との連結部のすぐ上流にトロンビンというプロテアーゼの認識配列 [
LVGRPS ] が
あって、そこで切り離すことができます。また、New England Biolabs で売られている
pMAL ベクターは約 42 kDa のマルトース結合タンパク(MBP)との融合タンパクを
作ることができます。この場合は MBP がマルトース(麦芽糖)と結合することを利用した
アフィニティークロマトグラフィーで精製できます。また精製後に MBP の下流にある
Factor Xa (これも血液凝固系で働くプロテアーゼです)認識部位
[ IEGR ] を切断
することによって、MBP 部分と切り離すことができます。
実験
1. 2-10 と同様に IPTG でタンパク発現を誘導した大腸菌をエッペンに
移し、遠心して集菌する。菌の沈殿を残し上清を捨てる。
グループで 1 サンプル。2. エッペンに バッファー B (a) を 1 ml 注ぎ、ボルテックスで混ぜる。
3. 10 ml 用チューブに移し、超音波破砕機で合計 3 分ほど sonication。
1 回に 1 分程度、氷冷しながら行う。30 秒ほど休んで、再び超音波・・・を
繰り返す。大腸菌が破壊されれば、液が透明になる。4. 溶菌液を新しいエッペンに移し、20‐30 分、冷却遠心する。
5. 遠心の間に、Ni2+‐NTA‐アガロースビーズをバッファー B で
平衡化する。
(1) ビーズを 50 μl エッペンにとり、まず、ビーズをけんだくしてある液を捨てる。
(2) 500 μl のバッファー B を加える。
(3) マイクロピペッタ−で混ぜた後、低速で遠心し、上清を捨てる。
(4) 再びバッファー B を 500 μl 加え、混ぜる。
(5) 低速で遠心し、上清を捨てる。(上清を捨てるのは、溶菌液の遠心が
終わる直前でよい)6. 溶菌液の遠心が終わったら、上清のうち 50 μl を別のエッペン
にとり、等量の 2 倍濃度 SDS-サンプルバッファーと混ぜる。
残りの上清を平衡化済みビーズの入ったエッペンに移す。
液はたくさん残してもいいので、沈殿を絶対にとらないように。7. 30 分ほど室温においておき、ときどきピペッタ−で混ぜる。
Ni2+‐NTA‐ビーズに His-タグ付きリコンビナントタンパクを吸着させる。8. 低速の遠心でビーズを沈殿させた後、上清を捨てる。
9. ビーズに バッファー C (b) を 1 ml 加え、混ぜる。
バッファー C はビーズの洗浄用。このバッファー中では His‐タグタンパクは
ビーズから離れない。ビーズの隙間などに(Ni2+-NTA に吸着したわけでも
ないのに)残っている不要なタンパクを除去する。10. 手回し遠心でビーズを沈殿させ、上清の 50 μl を別のエッペンに
移し、等量の 2 倍濃度 SDS‐サンプルバッファーを加える。
残りの上清を捨てる。11. ステップ 9 と 10 をもう 1 度繰り返す。
ただし、SDS-PAGE 用のサンプリングはしなくてよい。12. ビーズに バッファー E' (c) を 100 μl 加え、混ぜる。
バッファー E' には イミダゾール(d) が入っている。これによって、
His-タグタンパクと Ni2+-NTA との結合が阻害され、タンパクはビーズから
離れる。13. 室温で 5 分ほど置き、ときどき混ぜる。
14. 低速の遠心でビーズを沈殿させ、上清を新しいエッペンにとる。
このうち 50 μl を別のエッペンにとり、等量の 2 倍濃度の
SDS‐サンプルバッファーを加える。15. ステップ 12-14 をもう 1 度繰り返す。
ただし、SDS-PAGE 用のサンプリングはしなくてよい。16. SDS‐サンプルバッファーにとかした各サンプルを 80-90℃ で
5 分煮て、15-20 μl 程度を電気泳動する。
実験に使う試薬
8 M 尿素(b) バッファー C
100 mM リン酸二水素ナトリウム(NaH2PO4)
10 mM Tris-HCl (pH 8.0)
8 M 尿素(c) バッファー E’
100 mM リン酸二水素ナトリウム(NaH2PO4)
10 mM Tris-HCl (pH 6.3)
8 M 尿素(d) イミダゾール
100 mM リン酸二水素ナトリウム(NaH2PO4)
10 mM Tris-HCl (pH 8.0)
250 mM イミダゾール (下を参照)
図3 下図の左がヒスチジン、右がイミダゾール。図 2 と見比べよう。
イミダゾールが大量に持ちこまれると、このイミダゾールが Ni2+-NTA と結合し、
その結果、His‐タグタンパクがビーズから離れる。