変異タンパクの発現
pQE30-EGFP (mutants) の塩基配列と
SDS-PAGE
(2003 年 11 月 14 日)
pQE30 に組み込んだ変異型 EGFP cDNA の塩基配列を 11 月 13〜14 日の実験で調べて
みましたね。まずは、泳動の結果を載せます。残念ながら、一部のサンプルを除いてピークが
読み取れませんでした。必要なら画像をダウンロードしてレポートに使ってください。
WJ (室温培養したクローン) 
WR (池) 
WR (大?) 
YJ 
YR 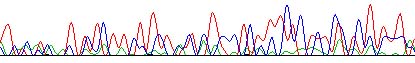
SR 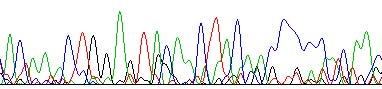
この SR は変異を入れた箇所の
付近です。「C」(青い線)が
連なっているところが変異導入
箇所ではないかと思います。
うまく配列が読めたのは以下の 2 サンプルです。
WJ (37℃ で培養した方): テキストのみ載せておきます。
TATGAGAGGATCGCATCACCATCACCATCACGGATCCGGTTATGGTCGTAAGAAGCGAC
GACAACGTCGTCGTGGTAAGGTACCCATGGTGAGCAAGGGCGAGGAGCTGTTCACCGGGG
TGGTGCCCATCCTGGTCGAGCTGGACGGCGACGTAAACGGCCACAAGTTCAGCGTGTCCG
GCGAGGGCGAGGGCGATGCCACCTACGGCAAGCTGACCCTGAAGTTCATCTGCACCACCG
GCAAGCTGCCCGTGCCCTGGCCCACCCTCGTGACCACCCTGACCTACGGCGTGCAGTGCT
TCAGCCGCTACCCCGACCACATGAAGCAGCACGACTTCTTCAAGTCCGCCATGCCCGAAG
GCTACGTCCAGGAGCGCACCATCTTCTTCAAGGACGACGGCAACTACAAGACCCGCGCCG
AGGTGAAGTTCGAGGGCGACACCCTGGTGAACCGCATCGAGCTGAAGGGCATCGACTTCA
AGGAGGACGGCAACATCCTGGGACACAAGCTGGAGTACAACTACAACAGCCACAACGTCT
ATATCATGGCCGACAAGCAGAAGAACGGCATCAAGGTGAACTTCAAGATCCGCCACAACA
TCGAGGACGGCAGCGTGCAGCTCGCCGACCACTACCAGCAGAA
SJ:
GTGGTAAGGTACCCATGGTGAGCAAGGGCGAGGAGCTGTTCACCGGGGTGGTGCCCATCC
TGGTCGAGCTGGACGGCGACGTAAACGGCCACAAGTTCAGCGTGTCCGGCGAGGGCGAGG
GCGATGCCACCTACGGCAAGCTGACCCTGAAGTTCATCTGCACCACCGGCAAGCTGCCCG
TGCCCTGGCCCACCCTCGTGACCACCCTGACCTACGGCGTGCAGTGCTTCAGCCGCTACC
CCGACCACATGAAGCAGCACGACTTCTTCAAGTCCGCCATGCCCGAAGGCTACGTCCAGG
AGCGCACCATCTTCTTCAAGGACGACGGCAACTACAAGACCCGCGCCGAGGTGAAGTTCG
AGGGCGACACCCTGGTGAACCGCATCGAGCTGAAGGGCATCGACTTCAAGGAGGACGGCA
ACATCCTGGGACACAAGCTGGAGTACAACTACAACCCCCACAACGTCTATATCATGGCCG
ACAAGCAGAAGAACGGCATCAAGGTGAACTTCAAGATCCGCCAC
SJ のピークはこんな感じです。
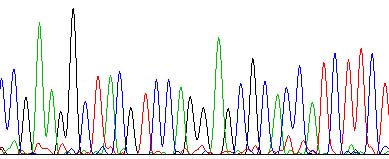
きれいに読めていますね。 pBluescript と比べて、pQE は、大腸菌細胞あたりのプラスミド
の個数が少なく、通常の miniprep では pBluescript ほどきれいに精製できない(ような
気がする)ので、塩基配列がきれいに読めないことが多いです。うまく読めなかった人は
原因をいろいろ考えてみてくださいね(そのためにピークの図を載せましたので、使って
ください)。
次は SDS-PAGE の結果です。
どれが誘導されたタンパクのバンドか、書かなくてもわかりますよね。分子量はアミノ酸配列
から計算した見積もり値と一致していましたか?タンパクが強く誘導されている大腸菌と
ほとんど誘導されていない大腸菌がいますね。原因を考えてみましょう。また、トランスイルミ
ネーターで大腸菌を光らせてみた結果を憶えていますか?(自分のグループに関係した分
くらいは憶えていますよね。)その結果と比較して検討してみてください。また、塩基配列を
読んだ結果とも比較してください。今回は何種類かのデータ、何種類かの大腸菌クローンの
比較をしなければいけません。複雑だし、一部データが揃っていない部分もあり、難しいだろう
と思いますが、よく頭の中を整理してレポートを書いてください。
ゲル No.1
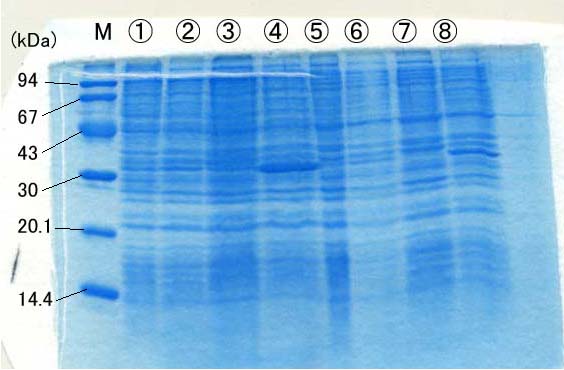
M: 分子量マーカー
1. YR(-) 2. YR(+) 3. SR(-) 4. SR(+) 5.
WJ-RT(-) 6. WJ-RT(+) 7. WJ37(-) 8. WJ37(+)
ゲル No.2
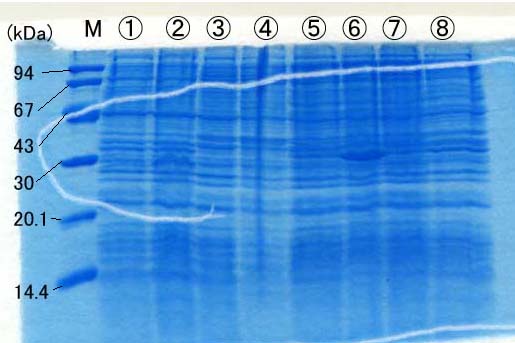
M: 分子量マーカー
1. YJ(-) 2. YJ(+) 3. SJ(-) 4. SJ(+) 5.
WR37(-) 6. WR37(+) 7. WR-RT(-) 8. WR-RT(+)
変異タンパク発現のページに戻る
遺伝子工学実験法のもくじのページに戻る








