海洋生命・分子工学実験 II
(7) リコンビナントタンパクの発現
はじ
めに
さて,いよいよです。組み込んだ cDNA
から EGFP
タンパクを作ってみましょう。ここでは,
転写も翻訳も大腸菌がやってくれます。でき
たタンパクが光るかどうか見てみましょう。
また,大腸菌の全タンパクを SDS-PAGE
によって分離し,EGFP タンパクのサイズが
期待どおりであるかどうかを確認しましょう。こういう方法で合成したタンパクは,精製して
活性を測定し,機能を解析することもできるし,正常に機能するタンパクを作ることが
できれば,それを医療などに応用できます。要するに,ここまでの一連の実験で学んだこと
は,決して実習のためだけの初心者専用の練習課題なのではなくて,直ちに第一線に
結びつく “役に立つ” 技術なのです。そのつもりで,ここまでの過程をよく頭と体に覚え込ま
せておきましょう。
知っておかなければな
らないこと
まずは図 1 を見てください。 (0) で一度見た pQE30 のマップですね。
図 1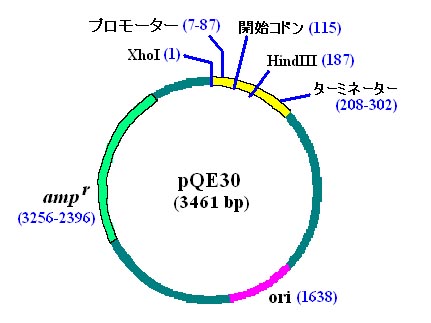
このプラスミドの multi-cloning
site の配列は図 2 です。これも (0) で見ましたね。
図 2 
この BamHI サイトと
HindIII サイトの
間に EGFP cDNA を組み込んだのでした。
理解していますか?大丈夫ですか? cDNA は,その 5' 側(翻訳されてできるタ
ンパクでいうと
N 末端側)を
BamHI 側に,3' 側(タ
ンパクでいうと C 末端側)
を HindIII 側に向けて組み込まれて
いるのですよね? cDNA の 5' 側にあたる側のプラスミドの配列には、promoter
(プロモーター)と
ribosome-binding site (リボゾーム結合部位)とがあります。図 2 では,プロモーター配列は
「-35 box」,
「-10 box」
と書かれています。(「分子遺伝学 C」を思い出そう!) プロモーターは
RNA 合成酵素の結合部位で, mRNA の転写はここから始まります。このプラスミドは T5
ファージ
の強力なプロモーターを持っています。また,プロモーター(RNA 合成酵素結合部位)のすぐ下流に
lac オペレーター(ラクトースオペロンのリプレッサー結
合部位の配列)が 2 つあります。
(これも「分子遺伝学 C」で教わったはず!) 念のために繰り返しますが,ラクトースオペロンで
は
リプレッサーと呼ばれるタンパク質がオペレーター配列に結合して転写を抑制しています。
ラクトース(乳糖)
を分解する酵素群の遺伝子が並ぶラクトースオペロンの場合,lacI
遺伝子が
作るリプレッサーがオペレーターに結合して転写を抑制しています。培地にラクトースを加えると
リプレッサーはラクトースと結合して立体構造が変化し,オペレーターから離れます。その結果,
抑制が解除してプロモーターからの転写が活性化し,ラクトース分解酵素群が生産されます。
ラクトースの消費によって培地中のラクトースが減少すると,リプレッサータンパクに結合する
ラクトースも少なくなります。ラクトースと離れたリプレッサーは再びオペレーターに結合し転写を
抑制します。そしてラクトース分解酵素群の生産は停止します。
・・・では、なぜpQE30 の T5 プロモーターにわざわざ lac オペレーターをつけたのでしょうか?
それは,組み込んだ cDNA からのタンパクの発現をコントロールするためです。
そもそも我々が
研究の対象にしようとするタンパクは,何らかの重要な生理活性を持っています。細胞の機能に
大きな影響を及ぼすそのようなタンパクは,往々にして大腸菌にとっては迷惑な(大腸菌の生育の
負担になったり害になるような)タンパクなわけです。(EGFP はほとんど無害ですが。。。)
そこで,常時発現させておくと大腸菌が生育せず,結局タンパクを得られないという結果になって
しまうおそれがあります。そこで,タンパクを発現させる実験の場合,まず組み込んだ遺伝子の発現
を抑制した状態で大腸菌をある程度増殖させ,その後に遺伝子発現を誘導するという方法が
とられます。たとえば,JM109 系統の大腸菌は F' エピゾームを持っています。F' の中には
lacIq 遺伝子があります。 lacIq
は野生型の lacI よりも強力なリプレッサーを作る変異遺伝子です。
この実験では,ラクトースのかわりにイソプロピル-β-D-ガラクトピラノシド
(IPTG)を用いて
転写を活性化します。IPTG(図 3)は,ラクトース同様にリプ
レッサーと結合し,オペレーターから
引き離しますが,ラクトースと違って分解されて減ることがないので,長時間安定に転写活性を
維持することができます。その結果,合成されるタンパクは大腸菌の細胞の中で最も量の多い
タンパクになります。
図 3 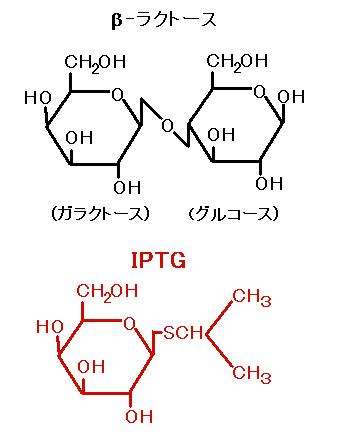
次に,pQE30
のプロモーターから
cDNA の連結部分まで(図 1),そして EGFP cDNA
の
N 末端の配列(図 4)をもう一度見てみましょう。このプラスミドには転写調節領域(プロモーターと
オペレーター),リボゾーム結合部位,開始コドンが含まれており,そ
の下流に制限酵素切断
部位を持っています。自分の目的の cDNA をこの制限酵素切断部位に組み込むだけで cDNA が
コードするタンパクを発現させることができるようにデザインされているのです。
図 4 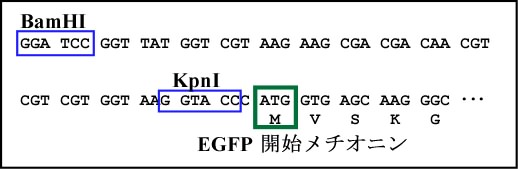
問題です!! 組み立てたプラスミドから作られるタンパ
クの N 末端部分のアミノ酸
配列を(N
末端から 35 アミノ酸)推定してください。できるかな?
いま発現させようとしているタンパクは,バクテリオファージのプロモーターに大腸菌のオペ
レーターとリボゾーム結合配列,さらに完全に人工的なヒスチジンリピート,そして EGFP の
翻訳領域が組み合わされてできた人工遺伝子の産物です。このように組換え DNA
(recombinant DNA) 技術によって作製された遺伝子の産物をリ
コンビナントタンパクと
いいます。
実験
(4) の実験で miniprep
に用いた大腸菌の試験管には,大腸菌が残っていますね。
その試験管を使います。
1. (Miniprep 用の大腸菌をとった後の)試験管に残った大腸菌に,1 mM IPTG
を
含む
LB/amp 培養液を 1 ml 加え,室温で振盪培養する。
同じ大腸菌を 2 本ずつ用意します。 1 本には IPTG 入りの培養液を,もう 1 本には
IPTG を含まない培養液を加えて,同じように培養しましょう。IPTG を入れたときと
入れなかったときの SDS-PAGE の泳動パターンを比較します。
2. 室温で実習終了直前まで培養した後,試験管の大腸菌をエッペンチューブに移し
遠心して菌を沈殿させる。
沈殿した大腸菌を直接トランスイルミネーターの上に置いて紫外線を当てて
みましょう。タンパクが発現していれば黄緑色に光るはずです。
3. 上清を捨て,SDS サンプルバッファーを加えてボルテックスで混ぜる。
SDS サンプルバッファーの組成については,専門海洋生命・分子工学基礎実験
(藤原
担当)あるいは海洋生命学実習 I
(湯浅先生担当)のレジュメを参照。
4. 80℃ から 90℃ 程度で 5 分煮て,菌を溶かす。
注: 菌が溶けると染色体 DNA が出てきて液がネバネバするので,ボルテックスをして
DNA を断片化し,液の粘性を下げておきます。この日の作業はここまでです。
次の時間に SDS-PAGE を行います。
5. SDS-PAGE によってタンパクを分離し,クマシーブルー染色をする。
SDS-PAGE に関する資料は別に配ります。
遺伝子工学的実験法の目次に戻る



