● RT-PCR
RT-PCR で遺伝子の発現量が比較解析できる原理は,以下の図を見てもらえば分かるはず。
moodle の講義動画も忘れずに。遺伝子 A の発現量は,①,②,③ の生体サンプルのなかで
① で一番高いとする。(実際の研究では,RT-PCRの結果から,はじめて分かることなのだけど,
実験の原理を理解するため,ここでは初めから分かっているとする)。
もしも,混ぜた直後(調製直後)の PCR 反応液のなかにある遺伝子 A の cDNA 鎖の本数を
数えることができたなら,下図の上向き矢印で示すような差があることが分かり,それで解析完了
なのだけど,それは現実的には不可能。
そのため,このRT-PCRでは,アガロースゲル電気泳動・エチジウムブロマイド染色によって
可視化(もしくは計測)できる量まで, 遺伝子 A の cDNA を PCR で増幅する。
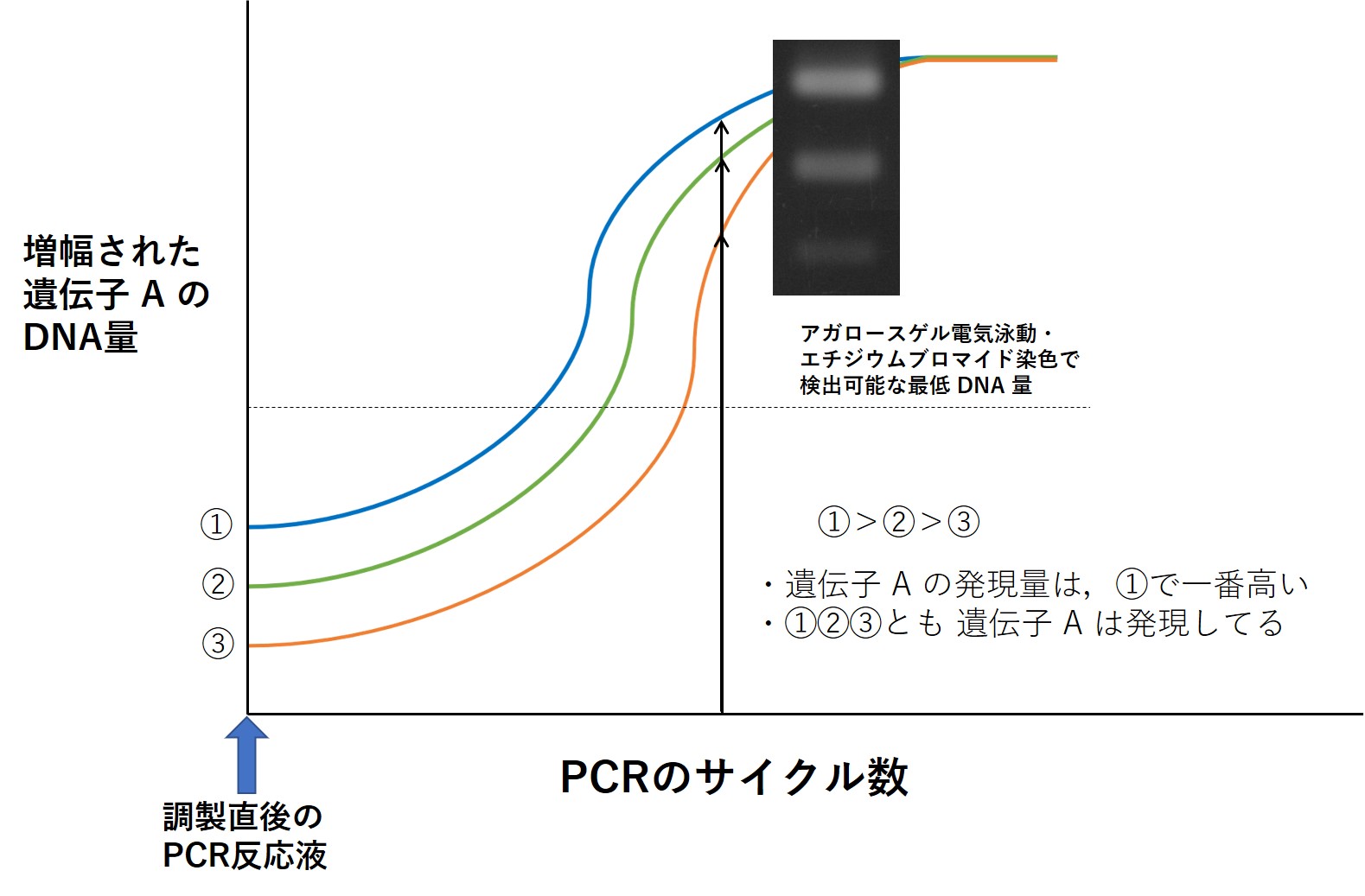
適当なサイクル数(実はここが肝心)で cDNA を PCR 増幅し,電気泳動,エチジウムブロマイド染色
により増幅された DNA を光るバンドとして検出する。増幅する前の PCR 反応液中の cDNA 量を反映した
バンドの光強度が得られる。↑図中の電気泳動写真(3本の白く光るバンド))を見れば,元のcDNA量を
反映したバンドの光強度になっていることが分かる。
★ 結果 ★
この実習では,16細胞期,胞胚期,原腸胚期,プルテウス幼生の異なる 4 つの発生段階から作製した cDNA を鋳型にして,
以下の 4 種類の遺伝子の発現を解析した。
GAPDH (遺伝子_内部標準)→ グリセルアルデヒド -3- リン酸脱水素酵素をコードする遺伝子
FGF(遺伝子 X) → Fibroblast growth factor: 線維芽細胞増殖因子をコードする遺伝子
Nodal(遺伝子 Y)→ TGF-beta ファミリーに属するシグナルタンパク質をコードする遺伝子
GATAa(遺伝子 Z)→ 転写因子をコードする遺伝子
★ 電気泳動写真の見方 ★
-GAPDH の発現量について-
この RT-PCR で GAPDH の cDNA を増幅したのは,GAPDH の発現量を比較したかったからではありません。
GAPDH の発現量を内在性コントロール(インターナルコントロール,内部標準,ノーマライザーなど呼び方は色々)
として利用するために増幅しました。 内在性コントロールには,細胞・組織の違いを問わず,一定量(高)発現している
遺伝子の発現量が使われます。厳密いうとサンプルによっては,GAPDH の他を選択する場合もあります,
内在性コントロールの説明も併せて,電気泳動の結果の見方を説明します。
ひとまず,比較するサンプル間で DNA のバンドの濃さ(光の強さ)に違いがあるならば,濃い方が薄い方に比べて発現量が
高いと解釈します。今回の実験なら,例えば,16 細胞期での発現量がプルテウス幼生期よりも高い,低い,同程度,,という
感じです。ただし,そのように見えるままに読み取ってよいのは,内在性コントロールの発現量に差がない場合だけです。逆転写反応
に使った total RNAは,各段階で同じ量としましたし,PCR 反応液に加えた cDNA の量も等しくしたのだから,条件はそろっている
はずです。ですが,吸光度測定による RNA 定量の不正確さや,逆転写反応でのチューブごとの微妙な条件の違いなどを想定しなけ
ればなりません。そういった違いを考慮して,必要ならば,結果を正しく補正します。。,
補正する場合の実際の操作は,まず,内在性コントロールの発現量を分母にとり,調べたい遺伝子の発現量を分子にして相対値を
算出します。その値を使って遺伝子の発現量を比較します。 この実習では,この数値化した結果を使った考察にもチャレンジしてみましょう。
電気泳動の写真では,多くの班で,内在性コントロール(GAPDH)の発現を示すバンドの濃さはほぼ同程度に見えます。
一方で,他の遺伝子のについてはバンドの濃さの差があるように見えます。
-アガロースゲル電気泳動-
全ての班で,一番左にマーカーが泳動してあります。その右隣から 8 レーンが各班のサンプルです。
8 レーンのうち,左から 4 つは内部標準として使う GAPDH の発現量を調べた結果で,残りの 4 つが
班ごとに違う遺伝子の発現量を示しています。。
1 班 (遺伝子 Y) と 2 班(遺伝子 X)

3 班 (遺伝子 Z) と 4 班 (遺伝子 Y)

5 班 (遺伝子 Z) と 6 班 (遺伝子 Y)
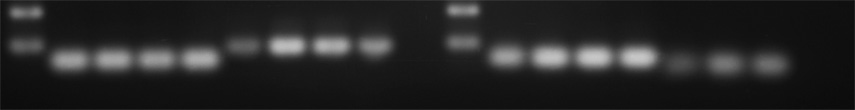
7 班 (遺伝子 X) と 8 班(遺伝子 X)

-電気泳動像の数値化-
この実習では,砂長の作業として,ImageJ というソフトウェアを使用して,電気泳動のバンドの光強度を
数値化しました。その結果をエクセルファイルにまとめたので,以下のデータを使って下さい。エクセルも pdf も
同じデータなので,使いやすい方を使ってください。
数値化エクセルデータはこちら
数値化 pdf データはこちら
★ レポート作成のポイント
まず,自分たちの解析した遺伝子の名前は,レポートでは,正式名称にして書いてください。(X,Y,Z のままではダメ)
電気泳動の結果と数値化データを参考にして,まずは自分たちが調べた遺伝子(X or Y or Z)の発現変動について
分かったことをまとめてください。さらに X or Y or Z 遺伝子のはたらきについて調べ,さらに,16細胞期,胞胚期,
原腸胚期,プルテウス幼生期がどのような段階だったかもよく思い出して,ムラサキウニの胚発生における X or Y or Z
遺伝子の役割について考察してみてください。また,ぜひ,他の班のデータについても考察してみてください。
各遺伝子のはたらきを自分で調べていくと,いろいろな壁にぶつかります。まず,ムラサキウニでのそれぞれの遺伝子の
はたらきについては,皆さんが直感的に欲しいと思う情報がそのまま書いてある HP や文献はおそらくありません。
関連する情報は,たくさん出てくると思います。他の動物種での情報だったり,遺伝子も似たような名前の遺伝子の情報が
あれこれ出てきたりします。そのどれを参考にしていいか,よく分からないと思います。それでも,そこにあまり悩まなくて
OK です。
大変だと思いますが,大切なのは,論理性です。話の筋道が通っていれば,多少「妄想」でも構いません。筋の通った妄想
なら評価します。遺伝子のはたらきも細かなことは正確に分からなくても,タンパク質のはたらきとして間違っていなければ
OK とします。例えば,転写因子が骨片の材料になる,,とかいうストーリーはさすがにダメですよね。また,その遺伝子の
はたらきを調べるためにはこんな実験をすればいい,,という考察も面白いと思います。
★ 課題3
RT-PCR による遺伝子発現解析では,PCR による増幅が含まれているため,mRNA を鋳型に合成された cDNA でなくとも,
鋳型となり得る DNA が PCR 反応液に混入すると,結果に大きな影響が出ます。こうした余計な増幅を除くための対策法を
調べてまとめなさい。今回の実習で,皆さんが実際に実践したかどうかは関係ありません。実習ではかなり手順を省略している
ので,砂長の説明や配布資料には載っていない対策がきっとあります。
★ 課題4(取組むかどうかは各人の任意とする)
課題3に書いた文章に関連して,仮に,鋳型としてはたらく DNA が完全に cDNA だけという PCR 反応液を準備できたとしても,
実際の細胞や組織における発現量を反映しない結果になってしまうことがある(実は,割と簡単にそういう状況に陥る)。
そこで,どうしてそうなってしまうのか(どういうときにそうなるのか)について説明し,さらに,それを解決する方法について
も記述してください。 課題 4 を無視しても,レポートに満点の評価はつくような採点基準になっています。知識の深化,,点数の
上積みを目指す人は,ぜひチャレンジ!
1) total RNA 抽出/RNA の品質チェック
2) 逆転写反応による cDNA 合成へ
TOPへ




